Pfadanalyse mit R lavaan
6. Cross-lagged panel design (Längsschnitt)
Arndt Regorz, Dipl. Kfm. & MSc. Psychologie, 24.04.2023
Dieses ist ein Tutorial über Cross-lagged-panel Designs (Längsschnittanalyse) in Pfadmodellen mit R lavaan.
Video
(Hinweis: Mit Anklicken des Videos wird ein Angebot des Anbieters YouTube genutzt.)
Warum Cross-Lagged-Panel-Modelle?
Eine spezielle Art von Pfadanalyse ist das Cross-Lagged Panel Modell (CLPM), das es ermöglicht, kausale Hypothesen zu Zusammenhängen zwischen Variablen in längsschnittlichen Daten zu untersuchen. Dabei kann man zwar in einem korrelativen Design eine Kausalität nicht abschließend beweisen. Jedoch ist eine notwendige Voraussetzung für eine kausale Wirkung temporal precedence, dass also die vermutete Ursache zeitlich vor der vermuteten Wirkung kommt. Und das kann man mit Längsschnittmodellen untersuchen, im Gegensatz zur Untersuchung in reinen Querschnittsmodellen.
Was sind Cross-Lagged-Panel-Modelle?
In einem Cross-Lagged-Panel-Modell werden zwei oder mehr Variablen zu mehreren Messzeitpunkten untersucht. Das ermöglicht die Untersuchung des zeitlichen Ablaufs von Änderungen in diesen Variablen und damit gewisse Rückschlüsse auf mögliche kausale Effekte. Das CLPM besteht aus zwei Hauptkomponenten: dem cross-lagged Pfad und dem autoregressiven Pfad.
Der cross-lagged Pfad repräsentiert die Beziehung zwischen unterschiedlichen Variablen über die Zeit. Er ermöglicht es, die Frage zu beantworten, ob die Werte einer Variable zu einem früheren Zeitpunkt die Werte einer anderen Variable zu einem späteren Zeitpunkt vorhersagen.
Der autoregressive Pfad repräsentiert die Beziehung einer Variable zu sich selbst über die Zeit. Er ermöglicht es, die Stabilität einer Variable über die Zeit zu prüfen, indem er die Vorhersagbarkeit der Werte einer Variable zu einem späteren Zeitpunkt basierend auf ihren Werten zu einem früheren Zeitpunkt untersucht.
Ein häufiges Einsatzgebiet von CLPM ist die Untersuchung von Mediation im Längsschnitt. Da einer Mediationshypothese zumindest theoretisch eine Kausalkette zugrunde liegt, bei der die UV den Mediator beeinflusst und der Mediator wiederum die AV, ist ein CLPM besonders geeignet, um diese vermutete zeitliche Abfolge zwischen diesen drei Variablen zu untersuchen. Dabei gibt es für Längsschnitt-Mediationen Modelle mit drei Messzeitpunkten, aber auch Modelle mit zwei Messzeitpunkten.
Hier wäre eine mögliche Modellgrafik eines einfachen Cross-Lagged-Panel-Modells mit nur zwei Variablen und zwei Messzeitpunkten.
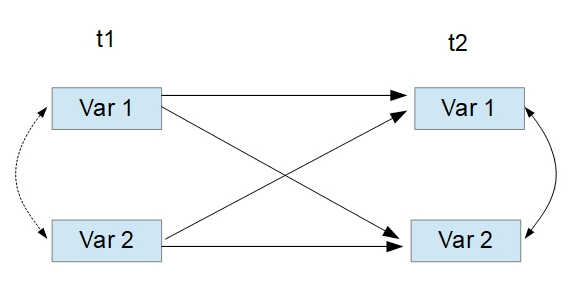
Cross-Lagged-Panel-Modell mit lavaan
Zunächst benötigen wir natürlich das lavaan Paket.
library(lavaan) head(meine_daten)
Bei der Modelldefinition eines CLPM wird jede Variable zu einem späteren Zeitpunkt durch die verschiedenen Variablen zu einem vorherigen Zeitpunkt vorhergesagt (durch sich selbst zum vorherigen Zeitpunkt und i.d.R. durch andere Variable(n)).
Dabei sollte man i.d.R. die Kovarianzen zwischen den verschiedenen Variablen eines Messzeitpunkts zulassen, da bei externen Störeinflüssen auf die Untersuchungseinheit zu irgendeinem Zeitpunkt nicht ausgeschlossen werden kann, dass dieser Störeinfluss auf mehrere Variablen wirkt.
Beispiel: Zu einem Messzeitpunkt ist eine Versuchsperson leicht erkrankt. Wenn sich diese Erkrankung auf mehrere der untersuchten Variablen auswirkt, ist damit zu rechnen, dass diese Variablen (bzw. technisch genauer: deren Disturbances) miteinander korrelieren. Wenn man diese Korrelation nicht bei der Schätzung berücksichtigt, kann ein schlechter Modellfit mit verzerrten Parameterschätzungen resultieren.
# Cross-Lagged-Panel-Modell
mein_modell <- '
# Gerichtete Pfade
Var1_t2 ~ AR11 * Var1_t1 + CL21 * Var2_t1
Var2_t2 ~ AR22 * Var2_t1 + CL12 * Var1_t1
# Kovarianzen
Var1_t2 ~~ Var2_t2
'
model_fit <- sem(data = meine_daten, model = mein_modell)
summary(model_fit, fit.measures = TRUE)
Natürlich darf man die Ergebnisse eines CLPM, wie jedes anderen Pfadmodells, nur bei einem akzeptablen Modellfit inhaltlich interpretieren. Für Zwecke dieses Beispiels gehe ich darauf nicht näher ein, siehe dazu das Grundlagentutorial zur Pfadanalyse mit lavaan.
# Visualisierung mit tidySEM
library(tidySEM)
pfad_layout <- get_layout("Var1_t1", "Var1_t2",
"Var2_t1", "Var2_t2",
rows = 2)
graph_sem(model = model_fit, layout = pfad_layout)
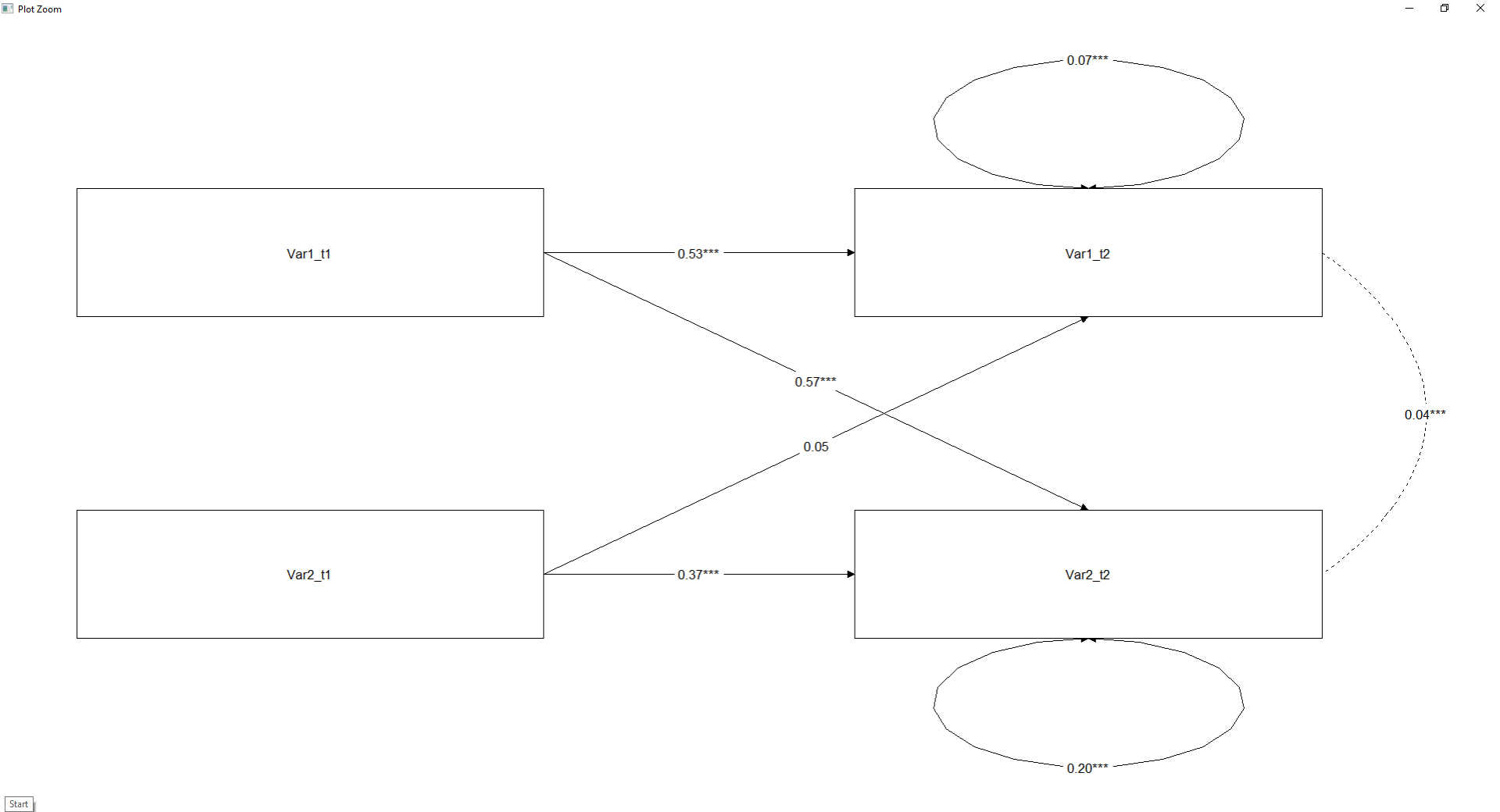
Hier im Beispiel sehen wir einen signifikanten Effekt von Variable 1 auf Variable 2, jedoch keinen signifikanten Effekt von Variable 2 auf Variable 1. Das spricht für eine kausale Wirkung von Variable 1 auf Variable 2 (ohne es aber damit endgültig beweisen zu können, s.o.).
Vergleich von zwei Cross-Lagged-Effekten
Wenn man wissen möchte, ob die Variable 1 stärker die Variable 2 beeinflusst oder umgekehrt, dann kann man das durch den Vergleich des obigen unbeschränkten (unconstrained) Modells mit einem zweiten Modell prüfen, indem beide Cross-Lagged-Effekte über eine Gleichheitsrestriktion gleichgesetzt werden.
# Signifikanztest auf Unterschied der beiden Cross-Lagged-Pfade
# Schätzung des Modells mit Gleichheitsrestriktion beider CL-Pfade
mein_modell2 <- '
# Gerichtete Pfade
Var1_t2 ~ AR11 * Var1_t1 + CL21 * Var2_t1
Var2_t2 ~ AR22 * Var2_t1 + CL12 * Var1_t1
# Kovarianzen
Var1_t2 ~~ Var2_t2
# Gleichheitsrestriktion Cross-Lagged-Pfade
CL21 == CL12
'
model_fit2 <- sem(data = meine_daten, model = mein_modell2)
summary(model_fit2)
Entscheidend ist jetzt der Likelihood-Ratio-Test (LR-Test, Likelihood-Quotienten-Test) zwischen diesen beiden Modellen. Die Nullhypothese ist, dass beide Kreuzpfade gleich stark sind. Wenn der Likelihood-Ratio-Test signifikant wird, kann man diese Nullhypothese verwerfen und hat gezeigt, dass beide Pfade unterschiedlich stark sind.
# Signifikanztest auf Unterschied von beschränktem und unbeschränktem Modell
lavTestLRT(model_fit, model_fit2)
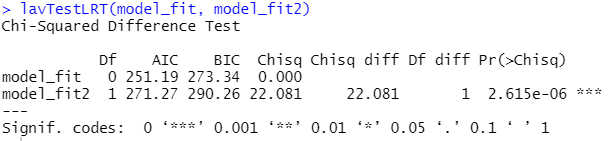
Hier sehen wir also, dass sich die beiden Cross-Lagged-Effekte signifikant voneinander unterscheiden.
Weitere Tutorials zur Pfadanalyse mit lavaan:
Pfadanalyse mit R / lavaan 1: Einführung
Pfadanalyse mit R / lavaan 2: Vergleich von zwei Pfaden
Pfadanalyse mit R / lavaan 3: Voraussetzungen und robuste Verfahren
Pfadanalyse mit R / lavaan 4: Moderation