Pfadanalyse mit R lavaan
1. Einführung
Arndt Regorz, Dipl. Kfm. & MSc. Psychologie, 02.02.2023
Mit lavaan, dem SEM-Modul von R, ist es einfach, eine Pfadanalyse durchzuführen - also eine Analyse, bei welcher der Zusammenhang zwischen gemessenen ("manifesten") Variablen betrachtet wird.
Inhalt
- Video-Tutorial
- Welche Schritte hat eine Pfadanalyse?
- Model specification
- Model identification
- Model estimation
- Model evaluation
- Model respecification
- Model interpretation
- Vollständiger R-Code zum Video
1. Einführungsvideo zur Pfadanalyse mit lavaan
(Hinweis: Mit Anklicken des Videos wird ein Angebot des Anbieters YouTube genutzt.)
2. Schritte einer Pfadanalyse
Die Durchführung einerPfadanalyse hat wie jedes SEM-Modell im wesentlichen 6 Schritte:
- Model specification
- Model identification
- Model estimation
- Model evaluation
- Model respecification
- Model interpretation
In der model specification wird das Modell aufgestellt. In der model identification wird sichergestellt, dass das Modell theoretisch schätzbar ist. In der model estimation wird das Pfadmodell geschätzt. In der model evaluation wird geprüft, ob das eigene Modell hinreichend gut zu den Daten passt. Falls nicht, wird in der model respecification das Modell geändert, damit es besser mit den Daten übereinstimmt. Und zum Schluss wird in der model interpretation das Modell interpretiert und mit Hilfe des Modells die eigenen Hypothesen beantwortet.
3. Model specification (Modellspezifizierung)
Bei der Modellspezifizierung wird das eigene theoretische Modell in lavaan-Code übersetzt, damit das Modell anschließend geschätzt werden kann.
Hier ist das Codebeispiel eines einfachen lavaan-Modells, das anschließend erläutert wird:
library(lavaan)
meine_daten <- read.csv("simulationsdaten_pfadanalyse.csv", header = TRUE)
Vor dem eigentlichen Code lädt man lavaan (vor dem erstmaligen Verwenden natürlich mit install.packages() installieren) und man lädt die Daten.
Hier ist das Modell, das wir schätzen möchten:
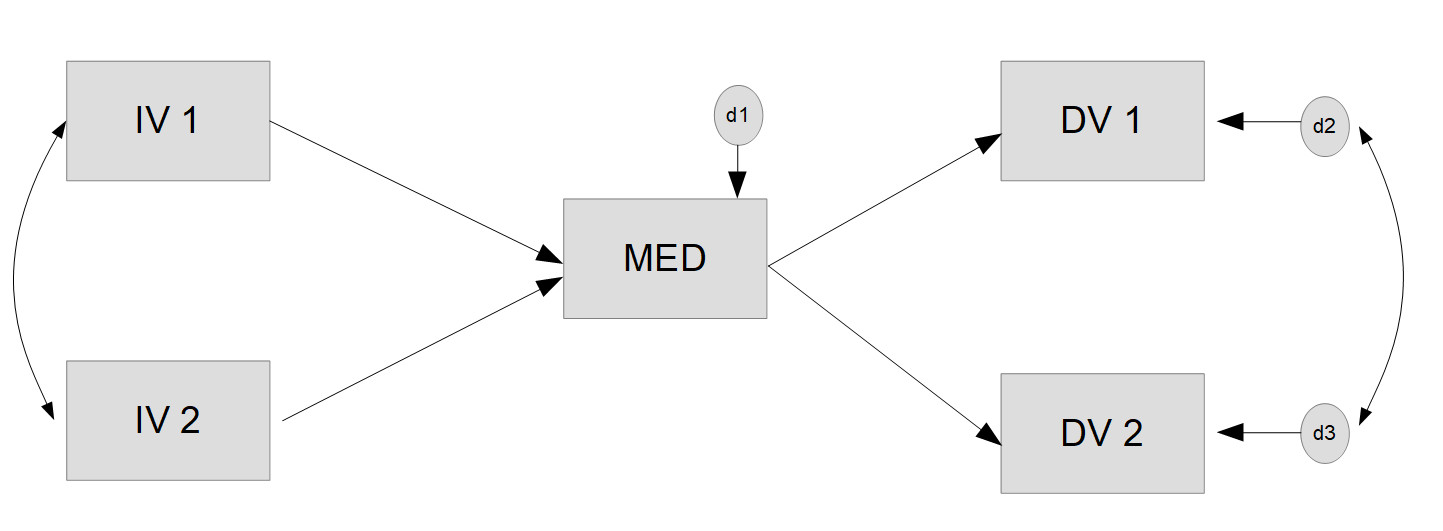
mein_modell <- '
# Gerichtete Effekte
MED ~ a1*IV1 + a2*IV2
DV1 ~ b1*MED
DV2 ~ b2*MED
# Kovarianzen
DV1 ~~ DV2
'
Die erste Zeile, MED ~ a1*IV1 + a2*IV2, bedeutet dabei: Die Variable MED wird durch die Variablen IV1 und IV2 vorhergesagt, das entspricht also eine multiplen Regression mit IV1 und IV2 als Prädiktoren und MED als Kriterium. Die Multiplikation der beiden Prädiktoren mit a1 und a2 sind Labels. Der Effekt von IV1 auf MED wird hier mit a1 benannt, der Effekt von IV2 auf MED mit a2; diese Namen kann man frei wählen und die (optionale) Verwendung derartiger Labels hat den Vorteil, dass man diese Labels später für weitergehende Berechnungen oder auch für Modellrestriktionen verwenden kann.
Die letzte Zeile, DV1 ~~ DV2, bedeutet, dass zwischen diesen beiden Variablen (oder technisch genauer: zwischen deren Disturbances/Fehlertermen) eine Korrelation geschätzt wird.
Neben diese beiden Befehlen, die in fast jedem Pfadmodell vorkommen, gibt es noch eine Reihe weiterer Befehle. Hier sind die wesentlichen Befehle noch einmal im Zusammenhang aufgelistet:
- AV ~ UV Gerichteter Effekt, UV sagt AV voraus
- AV1 ~~ AV2 Ungerichtete Kovarianz, AV1 und AV2 (bzw. deren Disturbances) korrelieren miteinander
- AV ~ 1 Intercept, es wird der Achsenabschnitt für die Variable AV geschätzt
- AV ~ a1 * UV Label, der Effekt von UV auf AV wird mit dem Namen a1 versehen
- ind := a1 * b1 Benutzerdefinierter Effekt, der Effekt ind ist hier das Produkt der beiden (irgendwo vorher definierten) Pfade mit den Labels a1 und b1
- a1 == a2 Restriktion, hier werden die beiden Pfade mit den Labels a1 und a2 exakt gleich geschätzt
4. Model identification (Modellidentifizierung)
Bevor man ein Modell schätzt (bzw. idealerweise bevor man überhaupt die Daten erhebt), sollte man sicherstellen, dass das eigene Modell überhaupt theoretisch schätzbar ist. Glücklicherweise ist das bei Pfadmodellen im Vergleich zu CFAs oder vollen SEMs in der Regel ein deutlich geringeres Problem.
Ich verwende dabei zwei einfache Faustregeln, um keine Probleme mit der Identifizierung zu haben:
1. Nur eine Verbindung zwischen zwei Variablen: Ich versuche, zwei Variablen nur mit maximal einer Verbindung zu versehen (Pfad von a auf b ODER Pfad von b auf a ODER Korrelation a und b).
2. Keine Kreisläufe: Ich verzichte auf Modelle mit Kreisläufen der Pfeile (also kein Modell, bei dem a auf b wirkt, b auf c, aber c dann wieder auf a).
Das sind nicht zwei notwendige Bedingungen - es kann gerade der Punkt 1. in seltenen Fällen auch mal durchbrochen werden müssen. Aber ich würde gerade als Anfängerin oder Anfänger einen großen Bogen um solche Modelle machen.
5. Model estimation (Modellschätzung)
Die Modellschätzung ist schnell gemacht:
model_fit <- sem(data = meine_daten, model = mein_modell)
Hier wird die sem-Funktion des lavaan-Moduls verwendet, ihr werden im einfachsten Fall der Dataframe übergeben, der ausgewertet werden soll, sowie die im Schritt model specification erstellte Modelldefinition.
6. Model evaluation (Modellevaluierung)
Für die Modellevaluation wird der Chi-Quadrat-Test als Modelltest betrachtet sowie die Fit-Indizes (z.B. CFI, RMSEA, SRMR). Beide zeigen den sog. global fit an, also wie gut das Modell insgesamt zu den Daten passt. Daneben sollte man noch den local fit betrachten, also ob es Teile des Modells gibt, in denen es Fit-Probleme gibt. Dazu vewrendet man sog. Modifikationsindizes.
summary(model_fit, fit.measures = TRUE)
mi <- modindices(model_fit)
mi[mi$mi > 10,]
Hier ist ein Auszug mit den wesentlichen Ergebnissen:
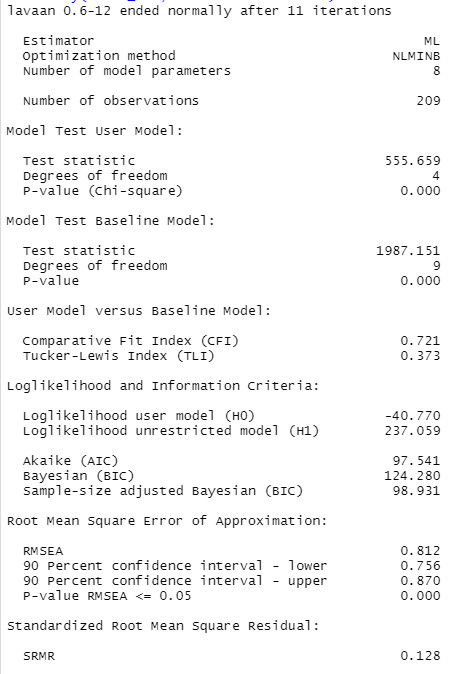
Wir sehen einen hochsignifikanten Modelltest (Chiquadrat 555.659 mit 4 Freheitsgraden). Auch die relevanten Fit-Indizes sind ziemlich schlecht, CFI = .721 (sollte über .95 sein), RMSEA = .812 (sollte je nach Quelle ca. unter .06 sein, wobei der RMSEA in Modellen mit wenigen Freiheitsgraden schlecht interpretierbar ist, siehe Probleme mit dem RMSEA ), SRMR = .128 (sollte unter .08 sein).
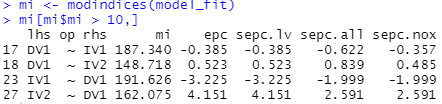
Auch die Modifikationsindizes (Spalte "mi") als Kennzeichen des Lokal-Fit zeigen, dass das Modell an einigen Stellen noch deutlich verbesserungsbedürftig ist.
7. Model respecification (Modellrespezifizierung)
Das das Modell noch nicht hinreichend zu den Daten passt, werden wir es jetzt respezifizieren. Dabei nutzen wir die o.g. Modifikationsindizes dazu, um weitere zu schätzende Effekte in das Modell einzufügen, die bisher noch vom Programm mit Null geschätzt werden (ein nicht spezifizierter Pfad wird im SEM-Modell als Null aufgefasst). Dabei sollte jeweils in einem Schritt nur i.d.R. eine einzige Veränderung vorgenommen werden, da eine Veränderung häufig gleich mehrere Modifikationsindizes zum Verschwinden bringen kann.
In der Spalte mi der Tabelle mit den Modifikationsindizes sehen wir eine ungefähre Schätzung, wie stark sich das Modell verbessern würde, wenn dieser Parameter (lhs: left hand side, op: Operator, rhs: right hand side) frei geschätzt wird. Wir sehen zwei ähnlich große Verbesserungsvorschläge: DV1 ~ IV1 und IV1 ~ DV1.
Allerdings sollte man nur theoretisch sinnvolle Veränderungen vornehmen. Ein Pfad von einer abhängigen Variable auf eine unabhängige Variable, wie bei IV1 ~ DV1, macht theoretisch keinen Sinn. Einen zusätzlichen direkten Effekt von der IV1 auf die DV1 hingegegen, wie bei DV1 ~ IV1, ist theoretisch gut begründbar. Insofern werden wir diese Änderung jetzt vornehmen und das damit geänderte Modell schätzen.
Hier ist das neue, respezifizierte Modell, das wir schätzen möchten (schon inkl. Labels und ohne die in der ersten Modellgrafik noch eingezeichneten Disturbances, die man in lavaan nicht explizit modellieren muss):
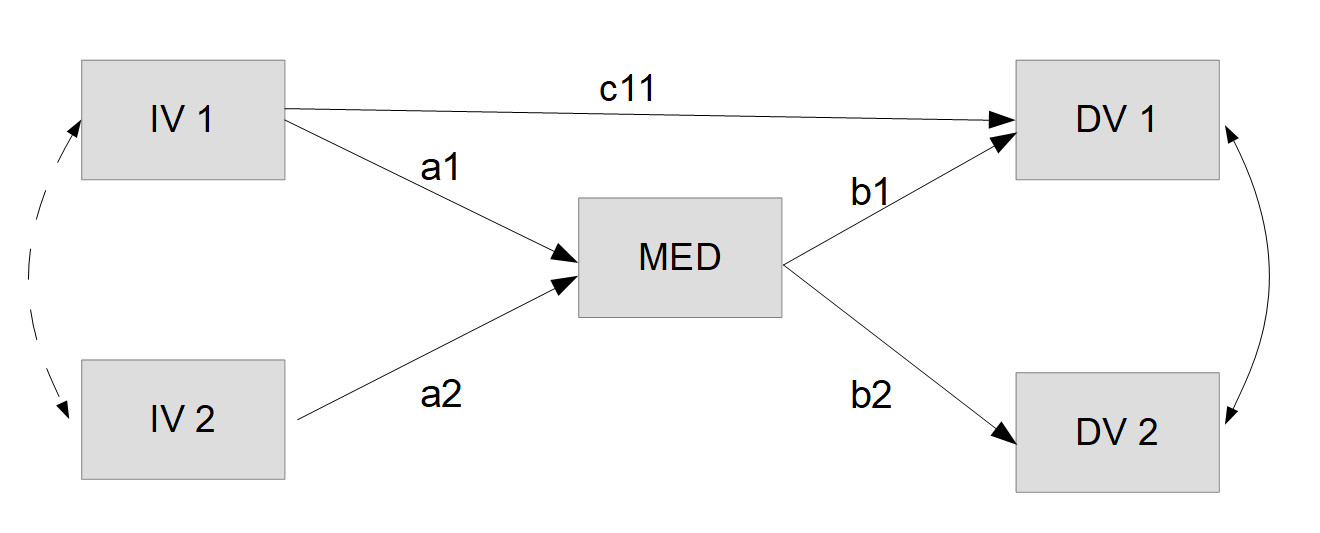
# Modell nach Modifikation
mein_modell2 <- '
# Gerichtete Effekte
MED ~ a1*IV1 + a2*IV2
DV1 ~ b1*MED + c11 * IV1
DV2 ~ b2*MED
# Kovarianzen
DV1 ~~ DV2
'
model_fit2 <- sem(data = meine_daten, model = mein_modell2)
summary(model_fit2, fit.measures = TRUE)
Hier ist wieder ein Auszug mit den wesentlichen Ergebnissen:
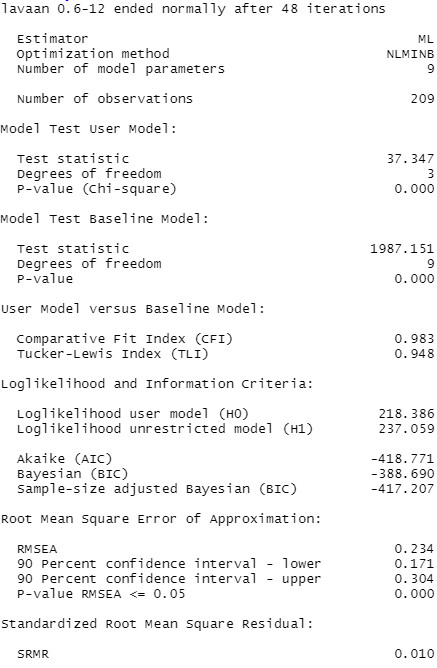
Wir sehen jetzt sehr gute Werte für CFI und SRMR (der Wert für den RMSEA ist aus dem o.g. Grund nicht sinnvoll interpretierbar bei hier nur 3 Freiheitsgraden). Dieses Modell ist also für uns akzeptabel und kann im nächsten Schritt von uns interpretiert werden. Vorher prüfen wir aber noch bei jeder Modellveränderung, ob sich das Modell auch signifikant verändert wird, mit dem Likelihood-Ratio-Test (LR-Test, Likelihood-Quotienten-Test, Chi-Quadrat-Differenzentest):
#Vergleich der beiden Modelle
lavTestLRT(model_fit, model_fit2)
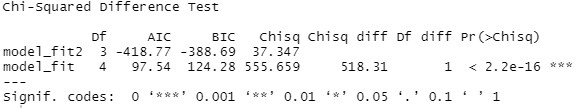
Wir erhalten eine sign. Verbesserung des Modells.
8. Model interpretation (Modellinterpretation)
Um die Ergebnisse zu interpretieren, würde ich noch zusätzlich standardisierte Effekte (betas) anfordern sowie Angaben zur Varianzaufklärung, R-Quadrat.
# Standardisiertes Ergebnis und R²
summary(model_fit2, fit.measures = TRUE, standardized = TRUE, rsquare = TRUE)
Interessant sind jetzt vor allem die "Regressions":
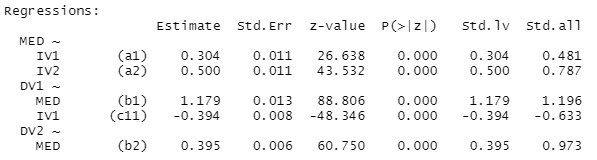
In der Spalte "Estimate" steht das jeweilige unstandardisierte Pfadgewicht (b), in der Spalte "Std.all" das standardisierte Pfadgewicht (beta). Der "z-value" ist die Teststatistik, die Spalte "P(>|z|)" zeigt an, ob der Effekt signifikant ist. Hier im Beispiel sind alle geschätzten Regressionspfade signifikant.
9. Vollständiger R-Code aus dem Video-Tutorial
Hier ist der gesamte Code aus dem Video-Tutorial:
library(lavaan)
meine_daten <- read.csv("simulationsdaten_pfadanalyse.csv", header = TRUE)
head(meine_daten)
mein_modell <- '
# Gerichtete Effekte
MED ~ a1*IV1 + a2*IV2
DV1 ~ b1*MED
DV2 ~ b2*MED
# Kovarianzen
DV1 ~~ DV2
'
model_fit <- sem(data = meine_daten, model = mein_modell)
summary(model_fit, fit.measures = TRUE)
mi <- modindices(model_fit)
mi[mi$mi > 10,]
# Modell nach Modifikation
mein_modell2 <- '
# Gerichtete Effekte
MED ~ a1*IV1 + a2*IV2
DV1 ~ b1*MED + c11 * IV1
DV2 ~ b2*MED
# Kovarianzen
DV1 ~~ DV2
'
model_fit2 <- sem(data = meine_daten, model = mein_modell2)
summary(model_fit2, fit.measures = TRUE)
#Vergleich der beiden Modelle
lavTestLRT(model_fit, model_fit2)
# Standardisiertes Ergebnis und R²
summary(model_fit2, fit.measures = TRUE, standardized = TRUE, rsquare = TRUE)
# Visualisierung mit tidySEM
library(tidySEM)
pfad_layout <- get_layout("IV1", "", "DV1",
"", "MED", "", "IV2", "", "DV2",rows = 3)
graph_sem(model = model_fit2, layout = pfad_layout)
Weitere Tutorials zur Pfadanalyse mit lavaan:
Pfadanalyse mit R / lavaan 2: Vergleich von zwei Pfaden
Pfadanalyse mit R / lavaan 3: Voraussetzungen und robuste Verfahren
Pfadanalyse mit R / lavaan 4: Moderation
Pfadanalyse mit R / lavaan 5: Mediation
Pfadanalyse mit R / lavaan 6: Cross-Lagged-Panel Modell
Pfadanalyse mit R / lavaan 7: Fehlende Werte
Pfadanalyse mit R / lavaan 8: Pfadanalyse mit Kovarianzmatrix
Pfadanalyse mit R / lavaan 9: Binäre und Ordinale endogene Variablen