R - Moderationsanalyse mit PROCESS Model 1
Testen einer Interaktion mit Hayes' PROCESS-Makro (Version 3.5 oder später) mit R
Arndt Regorz, Dipl. Kfm. & M.Sc. Psychologie, 24.04.2021
Seit langer Zeit ist PROCESS eine der bevorzugten Wege, um in SPSS eine Moderation (Interaktion) zu testen. Endlich, im Dezember 2020, gibt es das PROCESS-Makro auch für R. Dieses Tutorial zeigt Ihnen, wie Sie mit PROCESS in R / RStudio eine Moderationsanalyse durchführen können.
Inhalt
- Video-Tutorial
- Download von PROCESS
- Initialisieren des R-Codes
- Test eines Moderationsmodells
- Beispieloutput 1 – Moderation
- Zusätzliche Parameter
- Mein bevorzugter Moderations R-Code
- Beispieloutput 2 – Moderation mit Zusatzoptionen
- Weitere Informationen
1. YouTube-Video-Tutorial
(Hinweis: Mit Anklicken des Videos wird ein Angebot des Anbieters YouTube genutzt.)
2. Download von PROCESS
PROCESS für R ist keine R-Package, kann also nicht über install.packages() heruntergeladen werden (R-Packages mit dem Namensbestandteil "process" sind nicht das Original Process-Makro von Hayes, sondern mehr oder weniger gelungene Versuche von anderen, das nachzubauen.)
Hier kann man PROCESS für R herunterladen:
https://www.processmacro.org/download.html
Aktuell (April 2021) findet man dann im heruntergeladenen Zip-Ordner ein Verzeichnis mit dem Namen “PROCESS v3.5beta for R”. Dort ist eine R-Datei mit Namen "process.r" enthalten. Bitte berücksichtigen Sie, dass dies aktuell noch eine Beta-Version ist, die Fehler enthalten könnte.
3. Initialisieren des R-Codes
Da PROCESS keine R-Package ist, kann man die sonst von R gewohnten Kommandos install.packages() und library() dafür nicht nutzen. Als eine benutzerdefinierte Funktion muss man PROCESS installieren, indem man die o.g. Datei "process.r" in RStudio ausführt.
Wenn man process.r in RStudio öffnet, kann man es einfach ausführen. Das dauert eine oder zwei Minuten, und anschließend haben Sie eine neue R-Funktion zur Verfügung, process(). Mit dieser Funktion können Sie das PROCESS-Makro in Ihrer R-Umgegbung während der aktiven R-Session ausführen.
Wenn Sie diesen Code nicht jedes Mal neu aufrufen wollen, wenn Sie R neu starten (was ich jedoch tue, weil der Aufwand sich doch in Grenzen hält), dann empfiehlt Hayes, dass man den R Workspace speichert, nachdem man process.r hat laufen lassen.
4. Test eines Moderationsmodells
Der einfachste R/PROCESS-Code für eine Moderationsmodell wäre dieser:
process (data = my_data_frame, y = "my_DV", x = "my_IV", w ="my_MOD", model = 1)
In diesem Beispielcode habe ich die folgenden Variablennamen verwendet, die Sie mit den relevanten Namen aus Ihren Daten ersetzen müssten:
- my_data_frame: Mein Data-Frame mit den Daten, die ich für den Moderationstest nutzen möchte.
- my_DV: Der Name für die abhängige Variable in meinen Daten
- my_IV: Der Name für die unabhängige Variable in meinen Daten
- my_MOD: Der Namen für den Moderator in meinen Daten
Für Ihre Variablennamen sind einige wichtige Punkte zu bedenken:
- Der Variablenname muss in amerikanische Anführungszeichen (doppeltes Hochkomma) gesetzt werden.
- Bei Variablennamen unterscheidet R Klein- und Großbuchstaben - "moderator" ist z.B. in R eine andere Variable als "Moderator".
- Wenn Sie eine binäre Variable verwenden, dann darf diese nicht als Faktor in R definiert sein. Falls doch, müssten Sie diese zunächst in eine numerische Variable umwandeln.
Und zuletzt: Die Funktion "process" muss in Kleinbuchstaben geschrieben werden.
5. Beispiel-Output 1 – Moderation
Hier ist ein Beispiel des Outputs für den o.g. Code:
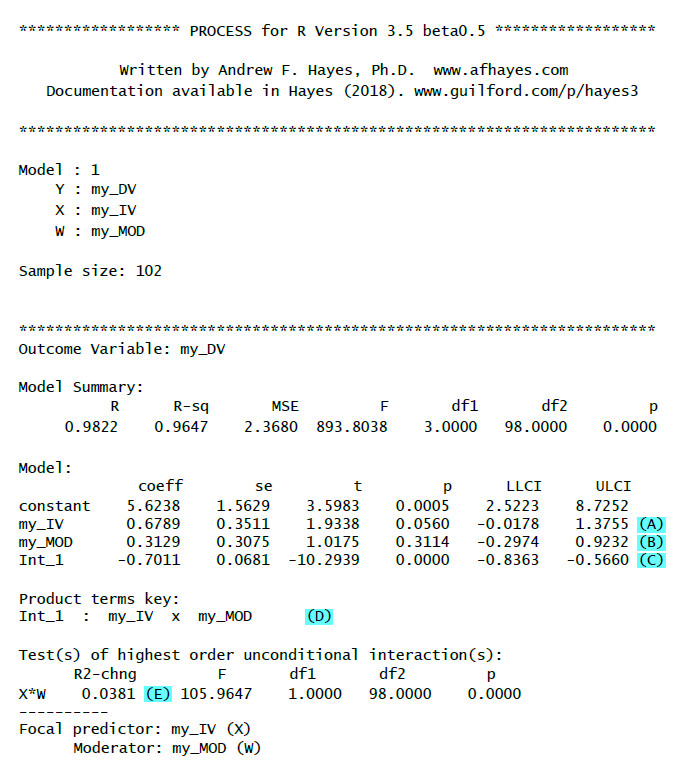
(A): Unstandardisiertes Regressionsgewicht (b) für die unabhängige Variable mit Test-Statistik, p-Wert und Konfidenzintervall
(B): Unstandardisiertes Regressionsgewicht (b) für den Moderator mit Test-Statistik, p-Wert und Konfidenzintervall
(C): Unstandardisiertes Regressionsgewicht (b) für die Interaktion mit Test-Statistik, p-Wert und Konfidenzintervall. Wenn es signifikant ist, liegt eine Moderation vor.
(D): Schlüssel für den Interaktionsterm (nur wichtig, wenn man ein Modell mit mehr als einer Interaktion hat)
(E): R2-chng: Effektstärke der Moderation (Wie viel zusätzliclhe Varianz erklärt die Interaktion über UV und Moderator hinaus?)
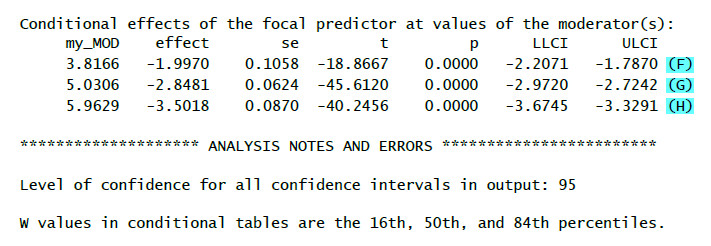
(F): Effekt (=b, zweite Spalte) der UV auf die AV für niedrige Werte des Moderators (16. Perzentil, erste Spalte) – Simple Slope
(G): Effekt (=b, zweite Spalte) der UV auf die AV für mittlere Werte des Moderators (50. Perzentil = Median, erste Spalte) – Simple Slope
(H): Effekt (=b, zweite Spalte) der UV auf die AV für hohe Werte des Moderators (84. Perzentil, erste Spalte) – Simple Slope
6. Zusätzliche Parameter
Auch wenn der obige Code Ihnen bereits einen Moderationstest liefert, möchten Sie in vielen Fällen zusätzliche Informationen haben. Nachfolgend finden Sie einige zusätzliche PROCESS-Parameter, die für Ihren Test, ob es eine Interaktion gibt, hilfreich sein könnten.
Mean Centering
In einer Moderationsanalyse ist die Interpretation der Regressionsgewichte einfacher, wenn Sie den Moderator (und vielleicht auch die unabhängige Variable) zentrieren. Wenn Sie den Parameter center auf 1 setzen, sind alle Variablen, die in Interaktionsterme eingehen (IV und Moderator), zentriert. Wenn Sie ihn stattdessen auf 2 setzen, werden nur kontinuierliche Variablen zentriert. Da die Zentrierung von binären Variablen die Interpretation der Ergebnisse erschwert, verwende ich nur diesen zweiten Parameterwert.
Beispiel:
center = 2
Ändern der Werte für Interaktionsdiagramm und Simple Slopes
Als Voreinstellung werden Interaktionsplots und Simple Slopes für den Median, das 16. und das 84. Perzentil ermittelt. Das können Sie ändern zu -1 SD, Mittelwert, + 1 SD (was ich bevorzuge), indem Sie den moments Parameter auf 1 setzen.
Beispiel:
moments = 1
Signifikanzregionen Johnson-Neyman
Um Signifikanzregionen nach Johnson-Neyman anzufordern, setzen Sie den jn Parameter auf 1 (sinnvoll bei kontinuierlichem Moderator).
Beispiel:
jn = 1
Diagramm für eine signifikante Interaktion^
Bei einer signifikanten Interaktion ist es sinnvoll, die Simple Slopes (Regressionsgeraden für verschiedene Werte des Moderators) graphisch darzustellen. Die dafür nötigen Daten kann man erzeugen, indem der plot Parameter auf 1 gesetzt wird.
Beispiel:
plot = 1
Für mich ist das in R (im Gegensatz zu SPSS) wenig hilfreich, weil ich dann ja immer noch R Code schreiben muss, um aus den Daten ein Diagramm zu machen. Daher würde ich dann bei einer signifikanten Interaktion eher auf eine R Package zugreifen, die wirklich direkt ein Interaktionsdiagramm mit Simple Slopes erzeugt: https://cran.r-project.org/web/packages/rockchalk/vignettes/rockchalk.pdf, siehe Kapitel 4.1 (Interaction in Linear Regression).
Hier wäre dann ein Code-Beispiel für Simple Slopes
(Zunächst muss man einmalig die rockchalk-Package installieren mit install.packages("rockchalk").)
library(rockchalk)
my_fit <- lm(my_DV ~ my_IV * my_MOD, data = my_data_frame)
summary(my_fit)
plotSlopes (my_fit, plotx ="my_IV" , modx = "my_MOD", modxVals = "std.dev." )
Berücksichtigung von Kontrollvariablen
Wenn Sie Kontrollvariablen/Kovariaten zusätzlich in Ihrem Modell berücksichtigen möchten, können Sie cov = ... nutzen. Bei einer einzelnen Kontrollvariable kann man diese einfach in die Formel einschließen. Bei mehr als einer Kontrollvariable müssen diese mit c(....) zusammen gebunden werden.
Beispiele:
cov = "age"
cov = c("age", "gender")
Bootstrapping
Wenn Sie robuste Konfidenzintervalle für Ihre Schätzungen erhalten möchten, können Sie den modelbt Parameter auf 1 setzen. Anderenfalls müssen Sie außerhalb von PROCESS Normalverteilung und Homoskedastizität prüfen, bevor Sie die Ergebnisse berichten können.
Beispiel:
modelbt = 1
Anzahl an Bootstrap-Stichproben
Die Voreinstellung beim Bootstrapping sind 5000 Stichproben. Diesen Wert können Sie ändern über den boot Parameter.
Beispiel:
boot = 10000
Bootstrapping - Startwert für den Zufallszahlengenerator
Bootstrapping enthält eine Zufallskomponente. Mit einem Zufallszahlengenerator werden Zufallsstichproben aus der Stichprobe gezogen. Das hat zur Konsequenz, dass bei jedem Mal, wenn Sie PROCESS mit den gleichen Daten ausführen, etwas andere Ergebnisse für die Konfidenzintervalle herauskommen. Das ist recht nervig, wenn man später die Ergebnisse zu Papier bringen möchte. Durch die Vorgabe eines (beliebigen ganzzahligen) Startwertes über den seed Parameter lässt sich das verhindern und jedes Mal kommt mit den gleichen Daten auch das Gleiche heraus.
Beispiel:
seed = 654321
7. Mein bevorzugter Moderations R-Code
Der folgende Beispielcode für eine Moderation mit zwei Kontrollvariablen (my_COV1, my_COV2) zeigt die Optionen, die ich meistens bei einem Test einer Moderationshypothese nutze.
process(data = my_data_frame, y = "my_DV",
x = "my_IV", w ="my_MOD",
model = 1, center = 2, moments = 1,
jn = 1, cov = c("my_COV1", "my_COV2"),
modelbt = 1, boot = 10000, seed = 654321)
8. Beispieloutput 2 – Moderation mit Zusatzoptionen
Hier ist ein Beispiel-Output mit den o.g. Zusatzoptionen. Kommentiert werden nur diejenigen Elemente, die nicht schon beim ersten Output erläutert worden sind.
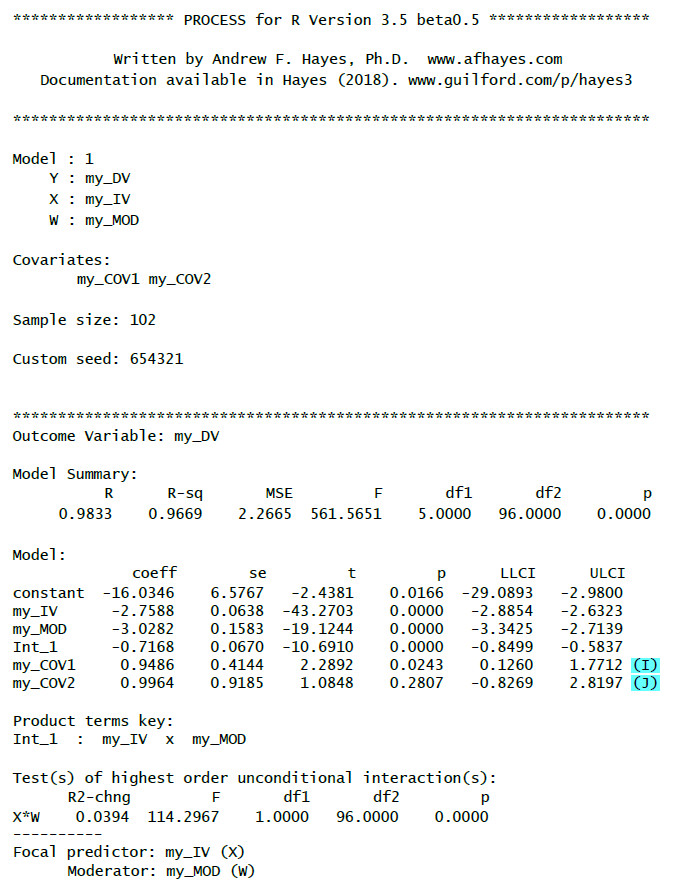
(I): Regressionsgewicht für die erste Kontrollvariable mit Test-Statistik, p-Wert und Konfidenzintervall
(J): Regressionsgewicht für die zweite Kontrollvariable mit Test-Statistik, p-Wert und Konfidenzintervall
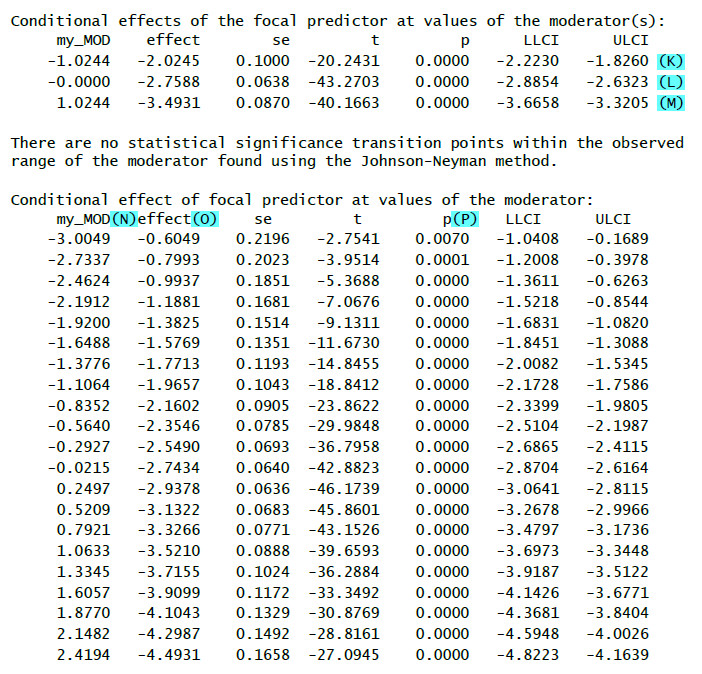
(K): Effekt (=b) der UV auf die AV für niedrige Werte des Moderators (1 SD unter Mittelwert) – Simple Slope
(L): Effekt (=b) der UV auf die AV für mittlere Werte des Moderators (arithmetischer Mittelwert) – Simple Slope
(M): Effekt (=b) der UV auf die AV für hohe Werte des Moderators (1 SD über Mittelwert) – Simple Slope
(N): Verschiedene Werte des Moderators
(O): Bedingter Effekt (b) der UV für diese Ausprägung des Moderators
(P): Signifikanztest für den bedingten Effekt
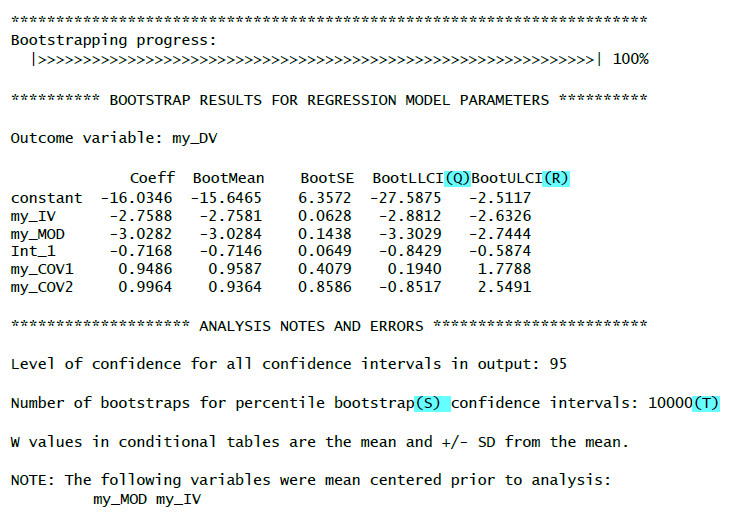
(Q): Untergrenze des Bootstrap-Konfidenzintervalls
(R): Obergrenze des Bootstrap-Konfidenzintervalls
Wenn die Null nicht im Konfidenzintervall liegt (also beide Grenzen positiv oder beide Grenzen negativ sind), dann zeigt das Konfidenzintervall einen signifikanten Effekt.
(S): Verwendete Bootstrappingmethode (Perzentil Bootstrap)
(T): Anzahl der Bootstrap-Stichproben (basierend auf der von Ihnen gewählten Anzahl)
9. Weitere Informationen
Wenn Sie mehr über die Theorie hinter der Moderationsanalys erfahren möchten, empfehle ich Andrew Hayes' exzellentes Buch:
“Introduction to Mediation, Moderation, and Conditional Process Analysis: A Regression-Based Approach”
http://www.afhayes.com/introduction-to-mediation-moderation-and-conditional-process-analysis.html
Weitere Tutorials zur Moderationsanalyse:
-
Grundlagen der moderierten Regression
Moderatoranalyse 1 -
Moderationsanalyse mit dem PROCESS-Makro für SPSS
Moderatoranalyse 2 -
Moderationsanalyse mit JASP
JASP Video-Tutorial Moderation -
Power/Stichprobenplanung für Moderationsanalysen
Moderatoranalyse G*Power