Finite Mixture Regression mit R
Moderatoranalyse ohne Moderator
Arndt Regorz, Dipl. Kfm. & MSc. Psychologie, 13.12.2023
Wenn Sie eine einfache oder multiple Regression durchgeführt haben, erhalten Sie für jeden Prädiktor in Ihrem Modell ein einziges Regressionsgewicht für alle Beobachtungen / Untersuchungseinheiten. Die Regression geht also implizit davon aus, dass die Zusammenhänge in Ihrer Population homogen sind, dass also für alle Untersuchungseinheiten die gleichen Zusammenhänge gelten.
Populationsheterogenität - Menschen sind nicht alle gleich
Doch das ist häufig keine realistische Annahme, insbesondere Menschen unterscheiden sich voneinander und damit auch in der Reaktion auf Versuchsbedingungen oder Umgebungsvariablen. Es ist also davon auszugehen, dass häufig Populationsheterogenität herrscht (= die Zusammenhänge sind nicht für alle Personen gleich). In so einem Fall sind die Ergebnisse der gewöhnlichen Regression lediglich Durchschnittswerte für die verschiedenen Effekte.
Ein Verfahren zum Umgang mit Populationsheterogenität kennen Sie schon: Die Moderationsanalyse. Allerdings funktioniert das nur bei beobachtbarer Populationsheterogenität, wenn Sie also schon einen konkreten Verdacht habe, welche Variable die Heterogenität Ihrer Daten erklärt und Sie diese auch bereits in Ihrem Datensatz erfasst haben.
Demgegenüber kann die Finite Mixture Regression (FMR) auch unbeobachtete Populationsheterogenität aufdecken. Wenn Sie signifikante Ergebnisse in Ihrer Regression erzielt haben, können Sie mit FMR anschließend explorativ prüfen, ob es Teilgruppen in Ihrem Datensatz gibt, die sich in den Regressionsgewichten (hinsichtlich Größe, Richtung, Signifikanz) voneinander unterscheiden.
Dieses Tutorial zeigt, wie Sie das einfach mit R umsetzen können. Vorher erläutere ich kurz zum besseren Verständnis von R Code und Auswertung einige Grundlagen zur FMR.
Wie FMR prinzipiell funktioniert
Es gibt einige Grundprinzipien, die man verstehen sollte, wenn man eine Finite Mixture Regression einsetzt:
- Latente Gruppen: FMR geht davon aus, dass es latente Gruppen (Teilpopulationen) in Ihren Daten gibt, die aber verborgen sind – wir haben also keine Gruppenvariable, an der wir die Gruppenzugehörigkeit ablesen können.
- Gruppenzugehörigkeit: Jeder Datensatz gehört zu einer dieser Gruppen, aber wir wissen nicht, zu welcher Gruppe. FMR findet das heraus.
- Separate Modelle: Jede Gruppe hat ihre eigene Art der Beziehung zwischen Variablen. Das heißt, die Regressionsgewichte unterscheiden sich zwischen den Gruppen, und möglicherweise auch deren Signifikanz. So hat ein Prädiktor in Gruppe A vielleicht einen viel stärkeren Einfluss als in Gruppe B, und ein zweiter Prädiktor hat in Gruppe A keinen signifikanten Einfluss, jedoch schon in Gruppe B.
- Gesamtmodell: Das endgültige Modell für alle Daten berücksichtigt all diese separaten Modelle, wobei der Beitrag jeder Gruppe basierend darauf gewichtet wird, wie wahrscheinlich es ist, dass ein Datensatz zu dieser Gruppe gehört. Es werden also die einzelnen Beobachtungen nicht eindeutig einer latenten Gruppe zugeordnet, sonder mit einen gewissen Wahrscheinlichkeit. So kann beispielsweise ein Fall mit 92% zu Gruppe A gehören, mit 0% zu Gruppe B und mit 8% zu Gruppe C.
Als Ergebnis einer Finite Mixture Regression kann man:
- Gruppen identifizieren: Wie viele und welche Arten von Gruppen sind in Ihren Daten vorhanden sind? Mit welchen Wahrscheinlichkeiten gehört ein Fall zu diesen Gruppen?
- Gruppen vergleichen: Wie unterscheiden sich die Beziehungen zwischen Variablen zwischen den Gruppen? Welche Prädiktoren spielen in welchen Gruppen eine Rolle?
- Vorhersagen treffen: Sobald Sie Ihr Modell haben, können Sie es verwenden, um Vorhersagen für neue Datensätze zu treffen. Diese Vorhersagen können je nach Zuordnung des neuen Datensatzes zu einer Gruppe unterschiedlich sein.
Praktische Umsetzung
Für die praktische Umsetzung gibt es verschiedene Teilschritte:
- Durchführen für verschiedene Anzahl an latenten Gruppen, denn wir wissen nicht, wie viele verschiedene Teilpopulationen in den Daten vorhanden sind.
- Bestimmung der optimalen Anzahl an latenten Gruppen, in erster Linie auf Basis von Informationskriterien.
- Vergleich der Regressionsergebnisse zwischen den Gruppen
- Extrahieren der Gruppenzugehörigkeit, d.h. für jede Versuchsperson/Beobachtung die latente Gruppe mit der höchsten Wahrscheinlichkeit
Darauf aufbauend (außerhalb der FMR) folgt die explorative Analyse, was die unterschiedlichen Gruppenzugehörigkeiten erklären kann, als Basis für eine spätere Moderatoranalyse. Man versucht, mit verschiedenen statistischen Verfahren, deskriptiven und schließenden (z.B. multinomiale logistische Regression), Erklärungen zu finden, warum welcher Fall in welche Gruppe zugeordnet worden ist.
Umsetzung mit R
Für eine FMR in R können wir das Package flexmix verwenden.
#install.packages("flexmix")
library(flexmix)
Demonstrieren werde ich die FMR mit einem Beispieldatensatz, der Teil des Packages ist, NregFix.
data("NregFix", package = "flexmix")
head(NregFix)
?NregFix
Aufruf mit fester Anzahl latenter Gruppen
Um die grundsätzliche Funktionalität zu zeigen, beginne ich mit einer Schätzung, bei der ich vorher die Anzahl der latenten Gruppen schon zu kennen glaube (was in der Praxis unrealistisch ist). Das erleichtert später das Verständnis für den Aufruf mit unbekannter Anzahl an Gruppen. Bei bekannter Gruppenzahl kann ich die Funktion flexmix() einsetzen, hier für 2 Gruppen:
fittedModel_2c <- flexmix(y ~ x2 + x1, k = 2, data = NregFix)
In der Funktion wird zunächst in der übliche Notation die Regressionsformel eingegeben. Dann folgt mit k die Anzahl der Gruppen und am Ende der verwendete Dataframe.
Wir betrachten zunächst die Übersicht über das Ergebnis.
summary(fittedModel_2c)
Call:
flexmix(formula = y ~ x2 + x1, data = NregFix, k = 2)
prior size post>0 ratio
Comp.1 0.536 97 194 0.500
Comp.2 0.464 103 122 0.844
'log Lik.' -524.1816 (df=9)
AIC: 1066.363 BIC: 1096.048
Zunächst wird die Formel wiederholt. Dann werden die extrahierten Gruppen dargestellt, hier wie vorgegeben zwei. Die a-priori Wahrscheinlichkeit, zu einer der Gruppen zugeordnet zu werden (prior) finde ich weniger interessant.
Das für mich wichtigste ist die Gruppengröße (size) – hier haben wir eine Gruppe mit 97 und eine Gruppe mit 103 Beobachtungen.
Dann folgt die Anzahl der Fälle, die eine Wahrscheinlichkeit von größer als 0 haben, in die jeweilige Gruppe eingeordnet zu werden (post>0). Für die erste Gruppe sind das 194 (von denen 97 am Ende in der Gruppe geblieben sind).
Und der letzte Wert ist das Verhältnis (ratio) zwischen Untersuchungseinheiten in einer Gruppe zu den Untersuchungseinheiten mit einer Wahrscheinlichkeit von größer 0 in dieser Gruppe, für Gruppe 1 sind das 97 / 194 = .500. Hier ist ein hoher Wert gut, weil er bedeutet, dass die meisten mit einer von 0 verschiedenen Wahrscheinlichkeit für die Gruppe auch am Ende dieser Gruppe zugeordnet worden sind. Insofern ist hier die Zuordnung zur Gruppe 2 mit einer ratio von .844 eindeutiger als die zur ersten Gruppe mit einer ratio von .500.
Hier ist noch wichtig, dass die Nummerierung der beiden Gruppen zufällig ist. Es wäre also genauso möglich, dass die beiden Zeilen vertauscht wären, wenn Sie die gleiche Auswertung mit den gleichen Daten aufrufen – es gibt also kein eindeutiges Kriterium, welche Gruppe die „Gruppe 1“ und welche Gruppe die „Gruppe 2“ ist.
Und zum Schluss sind noch verschiedene Gütemaße (logLik, AIC, BIC) für die Modellschätzung angegeben, die wir später nutzen können, um Schätzungen mit unterschiedlichen Anzahlen latenter Gruppen miteinander zu vergleichen (denn in der Realität wissen wir ja nicht, wie viele latente Gruppen wir in unseren Daten haben).
Was uns jetzt aber vor allem interessiert, das sind die Ergebnisse für die Regressionsgewichte in beiden Gruppen. Dafür nutzen wir die refit() Funktion.
summary(refit(fittedModel_2c))
$Comp.1
Estimate Std. Error z value Pr(>|z|)
(Intercept) -1.68352 0.60004 -2.8057 0.005021 **
x21 5.29089 0.86039 6.1494 7.777e-10 ***
x1 9.33768 0.39556 23.6065 < 2.2e-16 ***
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
$Comp.2
Estimate Std. Error z value Pr(>|z|)
(Intercept) 2.77595 0.13906 19.9628 <2e-16 ***
x21 5.19822 0.23221 22.3863 <2e-16 ***
x1 0.10394 0.11097 0.9366 0.349
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
Jetzt können wir die Regressionsgewichte für beide Gruppen vergleichen. Wir sehen, dass x1 nur in Gruppe 1 einen signifikanten Einfluss hat, nicht in Gruppe 2. Und wir sehen für x2, dass der Effekt in Gruppe 1 etwas größer ist als in Gruppe 2 (x21 wird für den Prädiktor x2 angegeben, weil es sich um einen binären Prädiktor handelt).
Wenn man die Parameter miteinander vergleichen will, geht es noch etwas besser mit der parameters() Funktion.
parameters(fittedModel_2c)
Comp.1 Comp.2
coef.(Intercept) -1.681309 2.7747970
coef.x21 5.293065 5.2074544
coef.x1 9.342845 0.1081012
sigma 4.206824 0.8942552
Jetzt sehen wir direkt nebeneinander die Parameterschätzungen für die beiden Gruppen. Gerade bei einer größeren Anzahl an Gruppen ist das so aus meiner Sicht übersichtlicher also die vorherige Darstellung, wenn auch ohne Angaben zur Signifikanz.
Aufruf mit unbekannter Anzahl latenter Gruppen
Normalerweise weiß man nicht, wie viele latente Gruppen es in den Daten gibt. Es kann sein, dass die Daten homogen sind hinsichtlich des Regressionsmodells (also nur eine Gruppe), es kann sein, dass es eine unbekannte Anzahl an latenten Gruppen (also mehr als nur eine) gibt mit unterschiedlichen Regressionsgewichten. Um das festzustellen, nutzen wir die stepFlexmix() Funktion. Diese Funktion probiert unterschiedliche Anzahlen an latenten Gruppen aus, so dass man anschließend auf Basis von Informationskriterien entscheiden kann, welche Gruppenzahl die beste ist.
fittedModel_1_5_c <- stepFlexmix(y ~ x2 + x1,
k = c(1,2,3,4,5),
nrep = 10,
data = NregFix)
Der Aufruf beginnt wieder mit der aus der normalen lm() Funktion bekannten Regressionsformel. Dann folgt die Anzahl der Gruppen, hier von 1 bis 5 (hier ist es vor allem wichtig, die 1 mit einzuschließen, weil es natürlich auch möglich ist, dass es keine Heterogenität in den Daten gibt und somit nur ein Regressionsmodell für alle Beobachtungen).
Dann kommen die Anzahl der Wiederholungen (nrep). Der Hintergrund für diesen Parameter ist, dass eine Schätzung mit dem EM Algorithmus nicht deterministisch ist, sondern auch eine Zufallskomponente enthält, so dass verschiedene Schätzungen zu unterschiedlichen Ergebnissen führen können. Der Wert von 10 bedeutet, dass für jede Anzahl an Gruppen die Schätzung 10 Mal durchgeführt wird und anschließend das Ergebnis mit dem besten Fit behalten wird. In der Praxis jenseits dieses einfachen Beispiels kann es sinnvoll sein, diesen Wert deutlich höher anzusetzen (z.B. 100), was dann aber auch entsprechend länger dauert. Denn je größer die Anzahl der Wiederholungen mit unterschiedlichen Startwerten, desto eher erhält man am Ende auch das globale Optimum bei den Parameterschätzungen und nicht lediglich ein lokales Optimum.
Und zuletzt kommt der Name des Dataframes.
Wenn wir diese Funktion starten, sehen wir zunächst nur Sterne.
1 : * * * * * * * * * *
2 : * * * * * * * * * *
3 : * * * * * * * * * *
4 : * * * * * * * * * *
5 : * * * * * * * * * *
Jeder Stern repräsentiert eine Schätzung. Es sind also für jede der von uns angegebenen Anzahlen latenter Gruppen (von 1 bis 5) jeweils 10 Schätzungen durchgeführt worden.
Jetzt betrachten wir eine Übersicht über die Ergebnisse für die fünf verschiedenen Anzahlen an Gruppen.
fittedModel_1_5_c
Wir erhalten als Resultat:
Call:
stepFlexmix(y ~ x2 + x1, data = NregFix, k = c(1, 2, 3, 4, 5), nrep = 10)
iter converged k k0 logLik AIC BIC ICL
1 2 TRUE 1 1 -641.7786 1291.5572 1304.7505 1304.750
2 24 TRUE 2 2 -524.1814 1066.3628 1096.0477 1135.484
3 26 TRUE 3 3 -451.7013 931.4027 977.5791 1006.617
4 44 TRUE 4 4 -446.6933 931.3865 994.0546 1033.226
5 43 TRUE 4 5 -446.6935 931.3871 994.0551 1033.227
Für jede von uns angegebene Zahl an Gruppen (1-5) haben wir eine Zeile, die das beste Schätzergebnis (aus in unserem Fall jeweils 10 Versuchen) pro Gruppenzahl wiedergibt.
Wichtig ist zum einen die Spalte „converged“. Nur wenn hier „TRUE“ steht, können die Ergebnisse interpretiert werden. Die vier Gütekriterien am Schluss der Tabelle dienen zum Vergleich der Gruppen. Sowohl nach BIC als auch nach ICL (Integrated Completed Likelihood Criterion, ein Entscheidungskriterium, dass im Gegensatz zum BIC speziell für Mixture Models entwickelt worden ist) ist die Lösung mit 3 Gruppen die beste, wohingegen nach AIC die Lösung mit 4 Gruppen minimal besser wäre als die Lösung mit 3. Ich würde jedoch generell BIC oder insbesondere ICL vorziehen, so dass dann eindeutig die 3-Gruppenlösung die beste ist.
Das können wir uns auch graphisch anzeigen lassen.
plot(fittedModel_1_5_c)
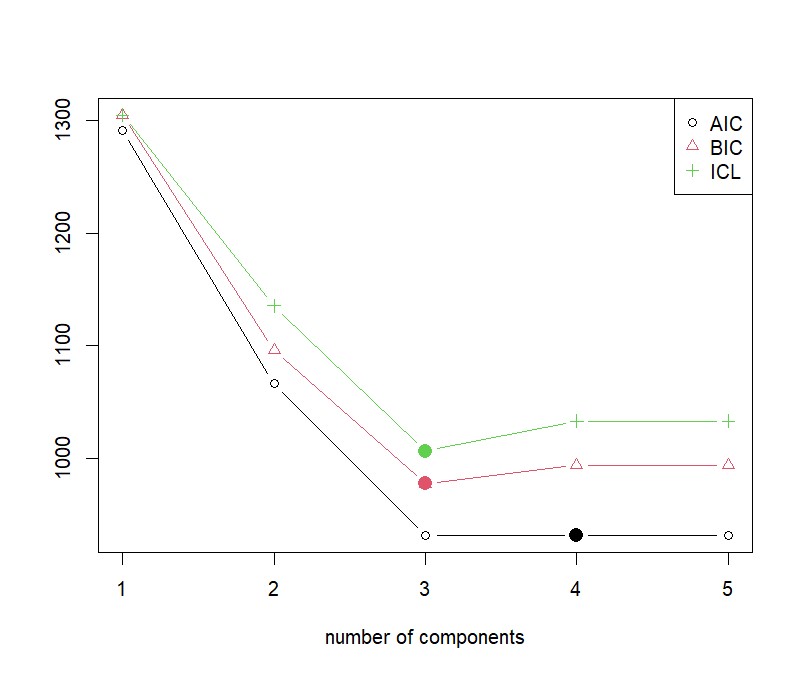
Auch hier sehen wir, dass die Lösung mit drei Gruppen die beste ist.
Daher wollen wir uns jetzt die Ergebnisse für diese Lösung genauer angesehen, um damit die Heterogenität in den Regressionsergebnissen zu verstehen.
Aus den Ergebnissen der stepFlexmix() Funktion extrahieren wir das Ergebnis für die 3-Gruppenlösung (mit which = 3).
best_model <- getModel(fittedModel_1_5_c, which = 3)
summary(best_model)
Als Ergebnis erhalte ich die nachfolgende Summary. Bitte beachten Sie, dass Sie mit den gleichen Daten zu minimal andere Ergebnisse für die drei Gruppen kommen können. Und zum anderen ist die Gruppenreihenfolge/-Benennung nicht eindeutig, so könnte bei Ihnen die Gruppe mit den 100 Untersuchungseinheiten z.B. auch unter Comp.2 oder Comp.3 auftauchen usw. (Das kann auch passieren, wenn Sie mehrmals die Funktion mit den gleichen Daten aufrufen.)
Call:
stepFlexmix(y ~ x2 + x1, data = NregFix, k = 3, nrep = 10)
prior size post>0 ratio
Comp.1 0.473 100 121 0.826
Comp.2 0.372 70 108 0.648
Comp.3 0.155 30 54 0.556
'log Lik.' -451.7014 (df=14)
AIC: 931.4027 BIC: 977.5792
Die Interpretation kennen Sie schon, siehe am Anfang des Tutorials bei der flexmix() Funktion.
Die Parameterschätzungen können wir auch genauso aufrufen, wie wir es oben mit der flexmix() Funktion getan hatten.
summary(refit(best_model))
parameters(best_model)
$Comp.1
Estimate Std. Error z value Pr(>|z|)
(Intercept) 2.842832 0.128790 22.0735 <2e-16 ***
x21 5.058363 0.208115 24.3056 <2e-16 ***
x1 0.098879 0.103268 0.9575 0.3383
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
$Comp.2
Estimate Std. Error z value Pr(>|z|)
(Intercept) 0.99110 0.17890 5.5398 3.028e-08 ***
x21 5.27447 0.25201 20.9295 < 2.2e-16 ***
x1 9.88960 0.11823 83.6461 < 2.2e-16 ***
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
$Comp.3
Estimate Std. Error z value Pr(>|z|)
(Intercept) -7.60234 0.26019 -29.218 < 2.2e-16 ***
x21 4.61886 0.38675 11.943 < 2.2e-16 ***
x1 9.96661 0.16379 60.849 < 2.2e-16 ***
---
Signif. Codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
> parameters(best_model)
Comp.1 Comp.2 Comp.3
coef.(Intercept) 2.8424246 0.9918487 -7.5999501
coef.x21 5.0612601 5.2734545 4.6158273
coef.x1 0.1000192 9.8894608 9.9670081
sigma 0.8932158 1.0494987 0.9831923
Wir sehen, dass wir zwei Gruppen haben, in denen beide Prädiktoren signifikant wurden (2 und 3) und eine Gruppe (1), bei der nur x2 signifikant wurde. Und Gruppen 2 und 3 unterscheiden sich deutlich hinsichtlich des Effekts von x2.
Wir können auch einen Rootogram für dieses Modell aufrufen:
plot(best_model)
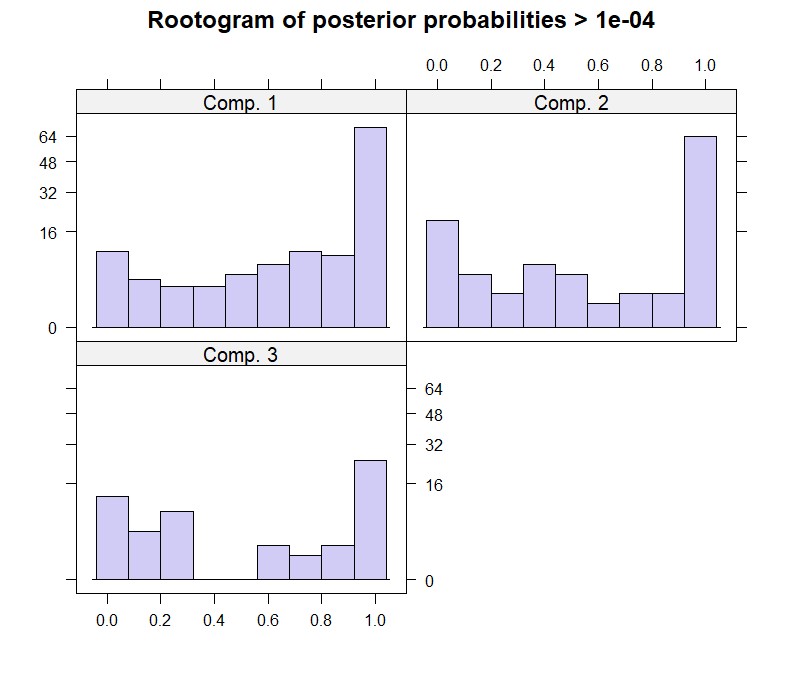
Beim Rootogram steht auf der x-Achse die Wahrscheinlichkeit der Zuordnung zu der jeweiligen Gruppe, auf der y-Achse die Quadratwurzel der Häufigkeit (es ist also sozusagen ein Histogramm, bei der auf der y-Achse nicht die Häufigkeit, sondern die Wurzel der Häufigkeit steht). Dabei werden nur diejenigen Beobachtungen ausgewertet, die für die jeweilige Gruppe eine Wahrscheinlichkeit größer als 0 (> 1e-04) aufweisen. Sie müssten sich also den Balken ganz links noch deutlich größer denken für die vollständigen Daten, wenn man auch die Beobachtungen mit einer Nullwahrscheinlichkeit mit in der Grafik hätte. Idealerweise sind die Balken ganz rechts jeweils deutlich größer als die Balken in der Mitte, was für eine relativ eindeutige Zuordnung von Beobachtungen zu den latenten Gruppen sprechen würde.
Die Wahrscheinlichkeiten für die Zuordnung einer konkreten Untersuchungseinheit zu einer der latenten Gruppen erhalten wir über die Anforderung der posterior-Wahrscheinlichkeiten.
posterior(best_model)
Die ersten Zeilen des Outputs sind:
[,1] [,2] [,3]
[1,] 1.778332e-01 6.787374e-18 8.221668e-01
[2,] 1.876444e-62 7.809698e-22 1.000000e+00
[3,] 7.225584e-14 1.000000e+00 1.566044e-21
[4,] 4.337200e-20 1.000000e+00 9.051944e-16
[5,] 9.907063e-01 9.293662e-03 4.014945e-09
Die erste Beobachtung ist also mit einer Wahrscheinlichkeit von .178 in Gruppe 1, von .000 in Gruppe 2 und von .822 in Gruppe 3.
Wenn wir eine Liste der jeweils wahrscheinlichsten Gruppenzuordnung haben möchten, fordern wir diese über die clusters() Funktion an.
clusters(best_model)
Wieder die ersten fünf Ergebnisse:
[1] 3 3 2 2 1
Die erste Beobachtung wird also in Gruppe 3 zugeordnet, ebenso die zweite, die dritte in Gruppe 2, usw. Sie können dies mit den Ergebnissen oben der posterior() Funktion abgleichen und werden sehen, dass hier jeweils die Gruppe mit der höchsten posterior-Wahrscheinlichkeit angegeben ist.
Diese Auswertung ist die Basis für weitere Untersuchungen hinsichtlich der Populationsheterogenität. Denn wenn Sie diese Spalte zu Ihrem Dataframe hinzufügen, können Sie beginnen, explorativ zu analysieren, was genau die Gruppenmitgliedschaft erklärt.
Sie können also beispielsweise alle anderen Variablen (außerhalb der für die Regression genutzten Variablen) getrennt nach dieser Gruppenzugehörigkeit auswerten, um die Merkmale zu finden, die die Gruppenzugehörigkeit vorhersagen können.
daten_cluster <- NregFix
daten_cluster$cluster <- factor(clusters(best_model))
head(daten_cluster)
x1 x2 w y class cluster
1 0.9243372 0 1 0.6758103 1 3
2 -0.3075028 1 1 -7.2930415 1 3
3 -0.6264132 0 0 -4.2606452 2 2
4 -0.6227993 0 0 -5.7047234 2 2
5 0.5807124 1 1 8.7911643 3 1
Jetzt kann man z.B. mit der describeBy() Funktion aus dem psych Modul sämtliche Daten des Dataframes getrennt für alle drei Gruppen anzeigen.
library(psych)
describeBy(daten_cluster, daten_cluster$cluster)
(Auf den Abdruck verzichte ich hier, da es im vorliegenden Datensatz neben den Regressionsvariablen nur die Variable w gibt, aber Sie haben normalerweise in Ihren Daten noch einiges an weiteren Variablen, z.B. soziodemographische Faktoren, usw.)
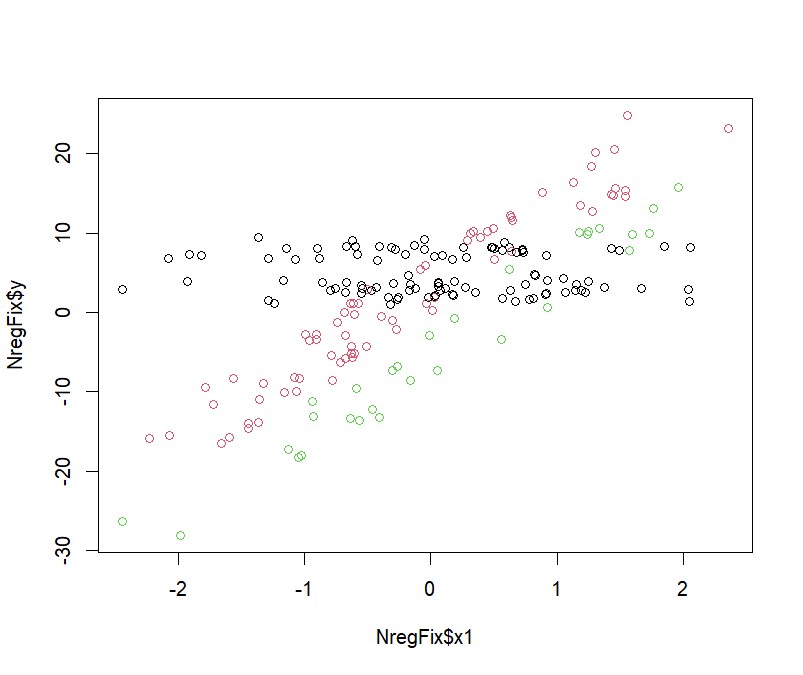
Und zuletzt können Sie sich noch ein Streudiagramm zwischen einer der UVs und der AV getrennt nach Gruppenzugehörigkeit anzeigen lassen. Das macht nur für kontinuierliche Prädiktoren Sinn, so dass es hier lediglich für den Prädiktor x1 aufgerufen wird. Bei mehreren kontinuierlichen Prädiktoren würde ich diese Grafik entsprechend mehrfach aufrufen (also den ersten Parameter entsprechend ändern).
plot(NregFix$x1, NregFix$y, col = clusters(best_model))
Literatur
Leisch, F. (n.d.). FlexMix: A general framework for finite mixture models and latent class regression in R. CRAN. Retrieved October 12th, 2023 from https://cran.r-project.org/web/packages/flexmix/vignettes/flexmix-intro.pdf