Strukturgleichungsmodelle mit R lavaan
1. Einführung
Arndt Regorz, Dipl. Kfm. & MSc. Psychologie, 30.10.2025
Dieses Tutorial zeigt die Grundlagen der linearen Strukturgleichungsmodellierung (SEM) mit lavaan, dem SEM-Modul von R.
(Hinweis: Mit Anklicken des Videos wird ein Angebot des Anbieters YouTube genutzt.)
Das Tutorial beruht auf dem sogenannten Two-Step-Approach. Dabei wird im Step 1 das Messmodell für die latenten Variablen überprüft und erst im Step 2 dann auch das Strukturmodell. Der Step 1 des Two-Step-Approach ist im Grunde eine konfirmatorische Faktorenanalyse (CFA), mit der geprüft wird, ob das vermutete Messmodell der latenten Variablen mit den Daten übereinstimmt. Erst wenn das der Fall ist, wird in Step 2 auch das Strukturmodell mit in die Analyse einbezogen. So können getrennt evtl. Fitprobleme in Messmodell und Strukturmodell identifiziert und ggf. bereinigt werden.
Step 1: Messmodell
Zunächst benötigen wir das lavaan Paket.
library(lavaan)
Der Datensatz, den wir verwenden, PoliticalDemocracy, ist Teil von lavaan, so dass Sie dieses Beispiel auch selbst nachvollziehen können.
head(PoliticalDemocracy)
Hier ist das Modell, das wir mit lavaan schätzen möchten:
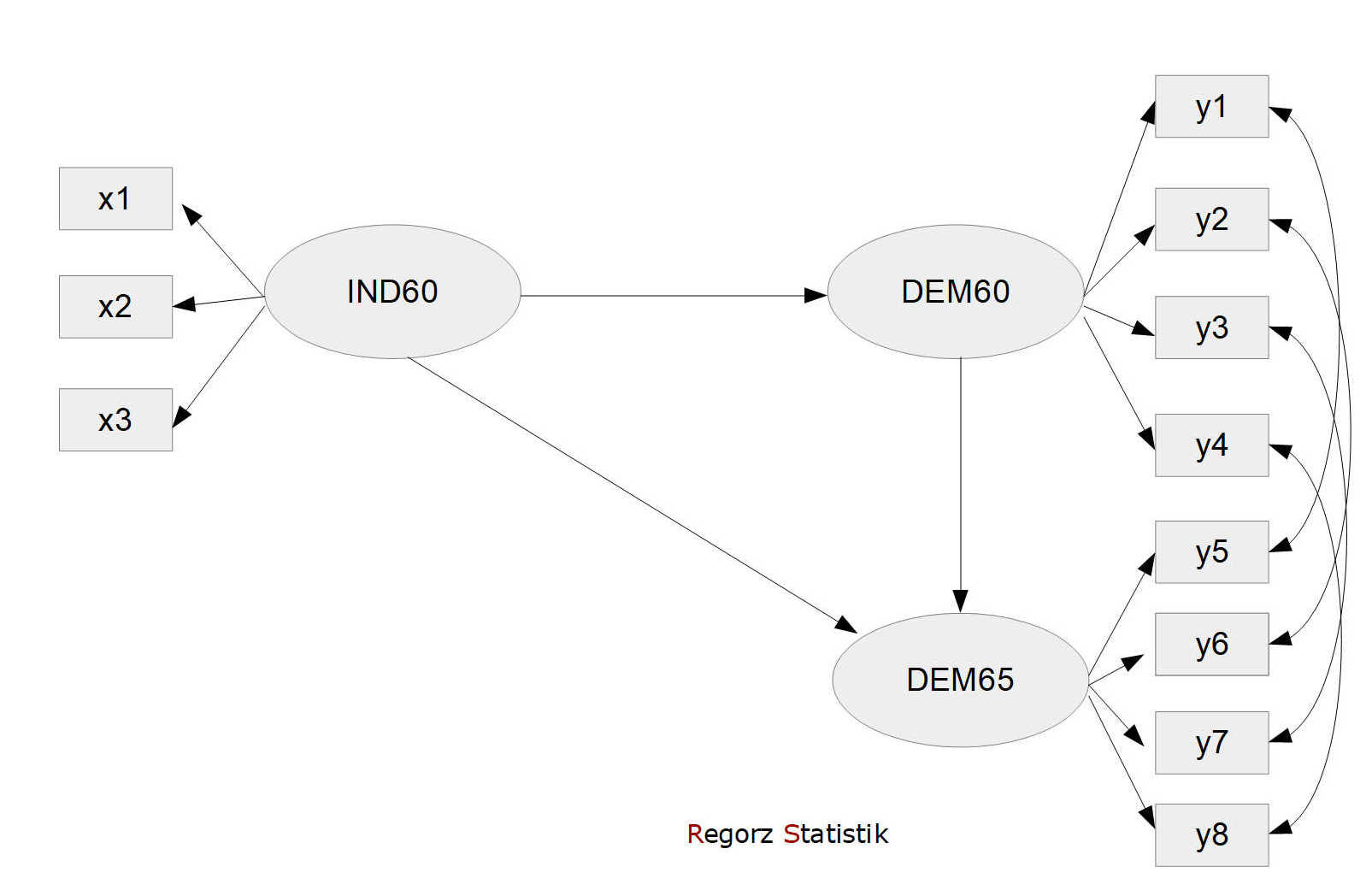
Zum Überprüfen des Messmodells führen wir eine CFA (konfirmatorische Faktorenanalyse) durch, bei der alle verwendeten Konstrukte frei miteinander kovariieren können. Alle dann evtl. bestehenden Fit-Probleme beruhen insofern ausschließlich auf des Spezifikation der Messmodelle.
In diesem Beispiel ist als Besonderheit zu berücksichtigen, dass ein Konstrukt (Demokratie) zu zwei Messzeitpunkten gemessen wurde, jeweils mit den gleichen Indikatorvariablen. In einem derartigen Fall solle man Fehlerkovarianzen zwischen den inhaltlich gleichen Items zu verschiedenen Zeitpunkten zulassen.
Für die Modellierung des Messmodells benötigen wir zwei Symbole:
Für die Faktorladungen nutzen wir das "=~"-Symbol.
Für Kovarianzen/Korrelationen (zwischen Faktoren oder zwischen Fehlertermen) nutzen wir das "~~"-Symbol.
Im Beispiel habe ich die Kovarianzen nur zwischen den Fehlertermen definiert, weil im vorliegenden Fall die Kovarianzen zwischen den latenten Faktoren / Konstrukten automatisch geschätzt werden, auch ohne dass man das explizit anfordert.
# Step 1: Überprüfung des Messmodells
messmodell <- '
# Faktorladungen
Industrialisierung1960 =~ x1 + x2 + x3
Demokratie1960 =~ y1 + y2 + y3 + y4
Demokratie1965 =~ y5 + y6 + y7 + y8
#Kovarianzen
y1 ~~ y5
y2 ~~ y6
y3 ~~ y7
y4 ~~ y8
'
Für die eigentliche Modellschätzung (model estimation) wird hier die sem-Funktion verwendet, weil diese auch später für die weitere Modellierung genutzt wird. Man hätte für diese CFA, denn das ist der erste Modellierungsschritt, aber auch die cfa-Funktion nutzen können.
model_fit1 <- sem(data = PoliticalDemocracy, model = messmodell)
Anschließend prüfen wir den Modell-Fit, weil nur bei einem akzeptablen Modell-Fit in einem weiteren Schritt das Strukturmodell hinzugenommen werden sollte.
summary(model_fit1, fit.measures = TRUE, standardized = TRUE)
Im Ergebnis erhalten wir hier einen guten Modellfit für das Messmodell:
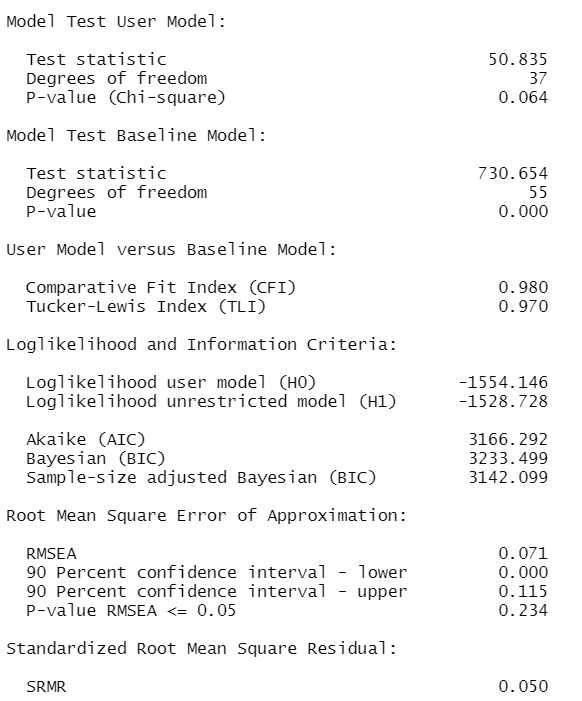
Der Chi-Quadrat-Test ("Model Test User Model") ist nicht signifikant, so dass wir die Nullhypothese eines perfekten Modellfits nicht verwerfen können. Der CFI ist mit .98 sehr gut, auch der SRMR ist mit .05 ziemlich gut. Der RMSEA ist mit .071 zwar etwas hoch, aber der Signifikanztest des RMSEA ist nicht signifikant. Insgesamt kann man hier sagen, dass das Modell gut zu unseren Daten passt.
Im Sinne des local-Fit sollten wir jedoch zusätzlich noch prüfen, ob auch alle einzelnen Teile des Modells passen. Das machen wir, indem wir Modifikationsindizes aufrufen:
mi <- modindices(model_fit1)
mi[mi$mi > 10,]
Es ergeben sich hier keine bedeutsamen Modellverbesserungs-Vorschläge, so dass wir das Messmodell in dieser Form akzeptieren können.
Die einzelnen Parameterschätzungen hinsichtlich der Korrelationen zwischen den drei latenten Variablen brauchen wir noch nicht zu betrachten, da es hier ja nur um einen Vorabschritt für die folgende Prüfung des Strukturmodells geht.
Falls uns der Fit jedoch noch nicht gereicht hätte, würden wir das Modell auf Basis der Modifikationsindizes weiter verbessern, was hier aus didaktischen Gründen dargestellt ist, auch wenn es mit den vorliegenden Daten nicht nötig ist.
# Einschub aus didaktischen Gründen: Wenn uns der Fit noch nicht reichen würde...
mi[mi$mi > 5,]
messmodell2 <- '
# Faktorladungen
Industrialisierung1960 =~ x1 + x2 + x3
Demokratie1960 =~ y1 + y2 + y3 + y4
Demokratie1965 =~ y5 + y6 + y7 + y8
#Kovarianzen
y1 ~~ y5
y2 ~~ y6
y3 ~~ y7
y4 ~~ y8
y6 ~~ y8 #zusätzlich zugelassene Fehlerkovarianz
'
model_fit1a <- sem(data = PoliticalDemocracy, model = messmodell2)
summary(model_fit1a, fit.measures = TRUE, standardized = TRUE)
Wenn man zwei genestete Modell miteinander vergleichen will, nutzt man dafür den Likelihood-Ratio-Test (Likelihood-Quotienten-Test), auch Chi-Quadrat-Differenzen-Test genannt:
lavTestLRT(model_fit1, model_fit1a)
# Ende Einschub aus didaktischen Gründen
2. Überprüfung des Strukturmodells
Nachdem wir uns vergewissert haben, dass das Messmodell zu den erhobenen Daten passt, können wir das Modell jetzt um den Strukturteil ergänzen, also um gerichtete Pfade zwischen den verschiedenen latenten Variablen.
Gerichtete Pfade, also Regressionspfade, modellieren wir dabei mit dem einfachen "~" Symbol - ganz genauso, wie wir es auch bei der Regression in R mit der lm()-Funktion machen würden.
# Step 2: Überprüfung des Strukturmodells
strukturmodell <- '
# Faktorladungen
Industrialisierung1960 =~ x1 + x2 + x3
Demokratie1960 =~ y1 + y2 + y3 + y4
Demokratie1965 =~ y5 + y6 + y7 + y8
# Strukturmodell/Regression
Demokratie1960 ~ Industrialisierung1960
Demokratie1965 ~ Demokratie1960 + Industrialisierung1960
#Kovarianzen
y1 ~~ y5
y2 ~~ y6
y3 ~~ y7
y4 ~~ y8
'
model_fit2 <- sem(data = PoliticalDemocracy, model = strukturmodell)
summary(model_fit2, fit.measures = TRUE, standardized = TRUE, rsquare = TRUE)
Bevor wir die Ergebnisse interpretieren können, müssen wir wieder den Modell-Fit betrachten, also den Chi-Quadrat-Test und die Fit-Indizes.
In diesem (Sonderfall) bei dem vorliegenden Modell erhalten wir die gleichen Werte zum Fit wie beim Messmodell. Das liegt daran, dass wir als Strukturmodell auf latenter Ebene eine einfache Mediation geschätzt haben, und dieses Strukturmodell hat 0 Freiheitsgrade, passt also immer perfekt zu den Daten.
Das wird aber bei komplexeren Modellen in der Regel nicht der Fall sein. Dann ist es relevant, wie viel sich der Modellfit vom Strukturmodell im Vergleich zum Messmodell verschlechtert. Bei einer deutlichen Verschlechterung würden wir anschließend wieder Modifikationsindizes anfordern, um festzustellen, an welchen Stellen das Strukturmodell ggf. noch nicht mit den Daten übereinstimmt.
mi <- modindices(model_fit2)
mi[mi$mi > 10,]
In unserem Beispiel haben wir jetzt ein akzeptables Modell und können nun die Ergebnisse interpretieren.
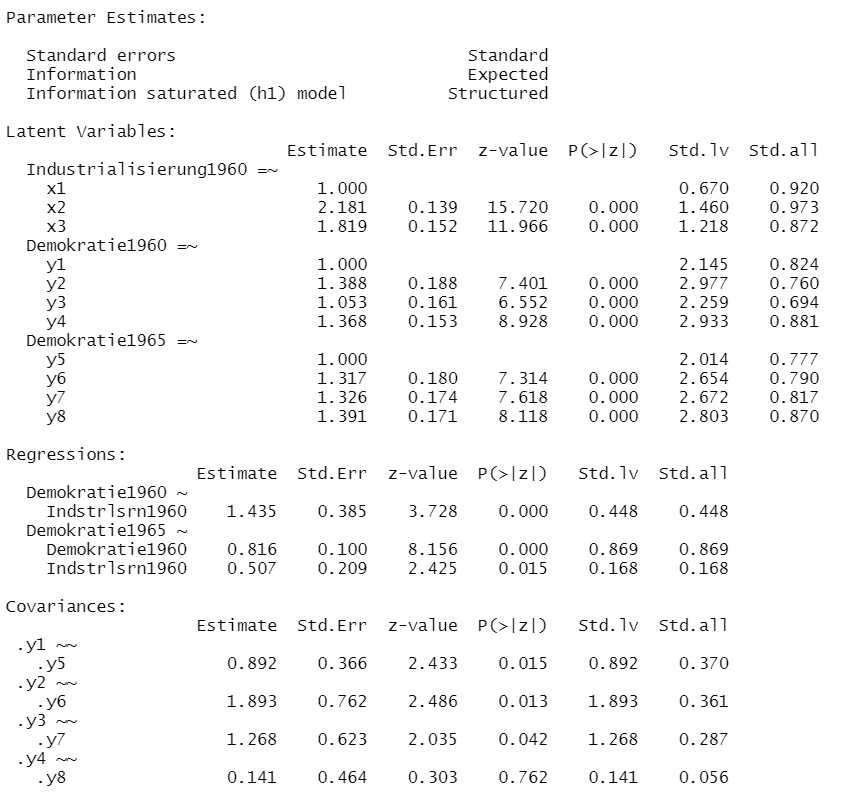
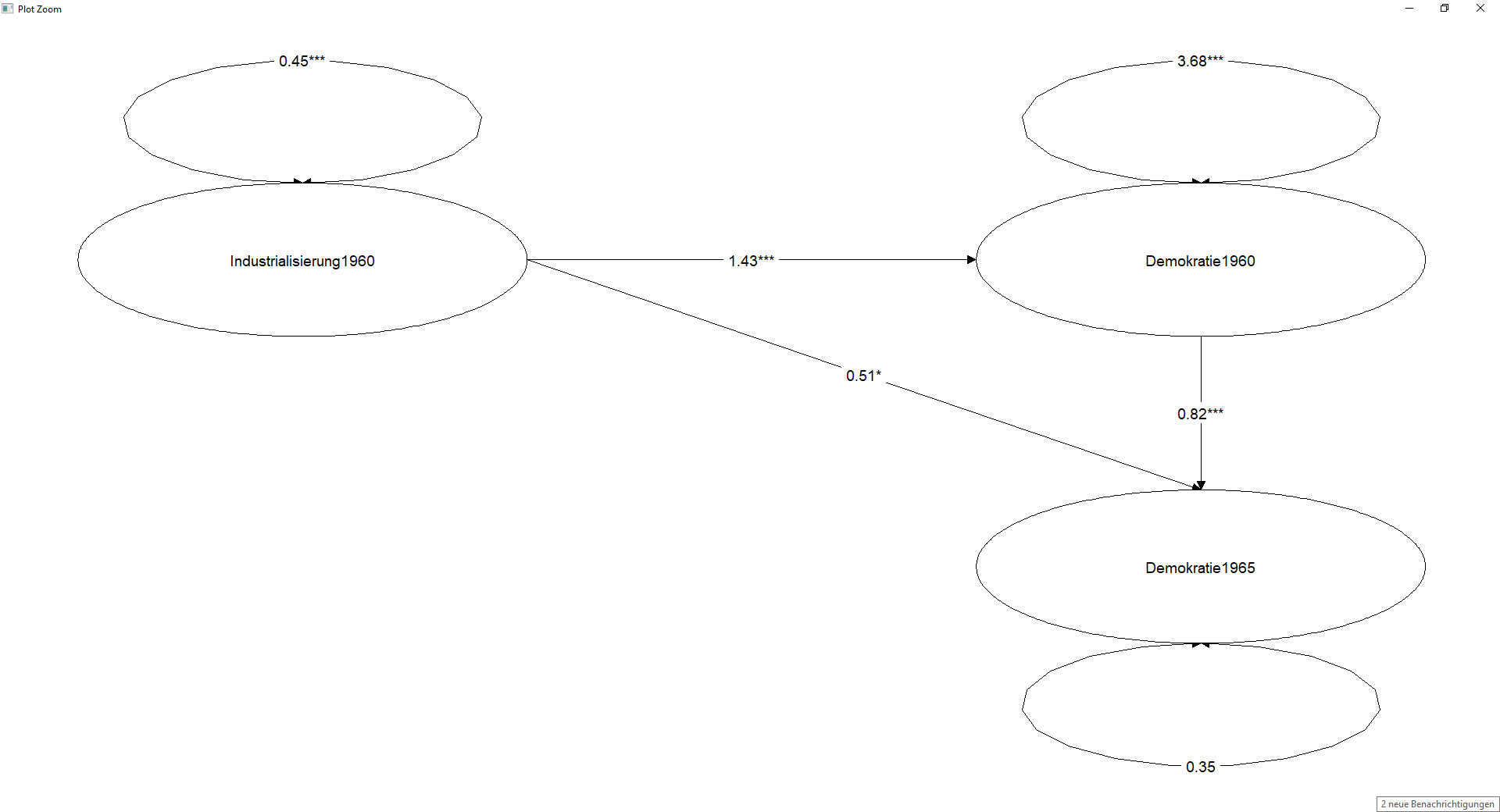
Primär von Interesse ist für uns bei einem SEM in aller Regel das Strukturmodell. Dieses finden wir unter der Überschrift "Regressions". Wir finden signifikante Effekte von Industrialisierung1960 auf Demokratie1960,
von Demokratie1960 auf Demokratie1965 und von Industrialisierung1960 auf Demokratie1965.
3. Modellvisualisierung
Zum Abschluss können wir die Ergebnisse der Modellschätzung noch von R visualisieren lassen. Für die graphische Darstellung von SEM-Modellen stehen in R verschiedene Packages zur Verfügung. Ich nutze gerne das tidySEM-Package.
# Visualisierung mit tidySEM
library(tidySEM)
pfad_layout <- get_layout("x1", "","", "", "y1",
"x2","Industrialisierung1960", "","", "y2",
"x3", "", "", "Demokratie1960", "y3",
"", "", "", "", "y4",
"","", "","","y5",
"","", "","Demokratie1965","y6",
"","", "","","y7",
"","", "","","y8",
rows = 8)
graph_sem(model = model_fit2, layout = pfad_layout)
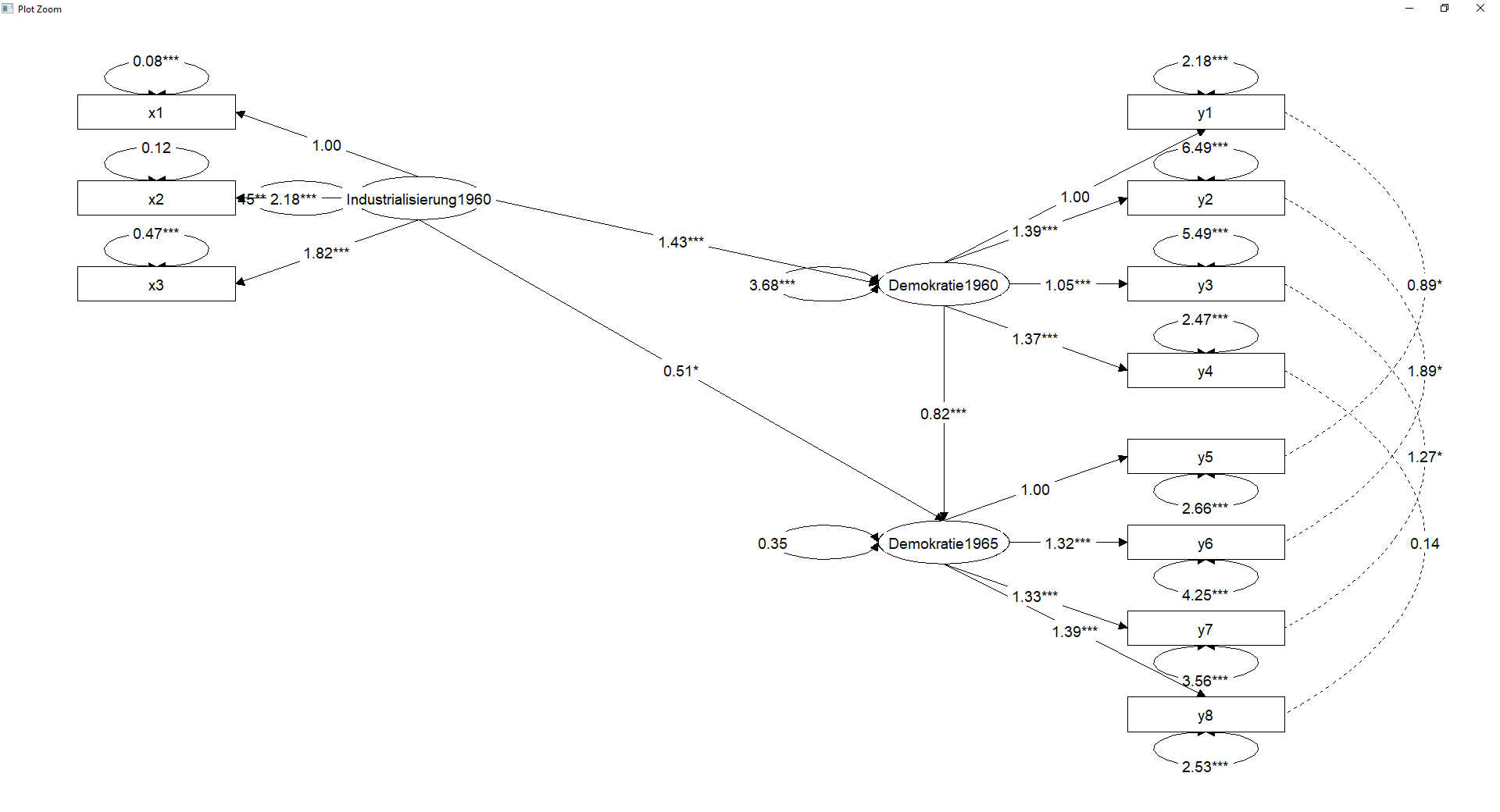
# Visualisierung ohne die Messmodelle, nur latente Variablen
pfad_layout2 <- get_layout("Industrialisierung1960", "Demokratie1960",
"","Demokratie1965",
rows = 2)
graph_sem(model = model_fit2, layout = pfad_layout2)
4. Literatur
Brown, T. A. (2015). Confirmatory factor analysis for applied research. Guilford publications.
Hu, L. T., & Bentler, P. M. (1999). Cutoff criteria for fit indexes in covariance structure analysis: Conventional criteria versus new alternatives. Structural Equation Modeling, 6(1), 1-55. https://doi.org/10.1080/10705519909540118
Kline, R.B. (2023). Principles and practice of structural equation modeling. Guildford publications.
Rosseel, Y. (n.d.). Lavaan tutorial. Retrieved December 16, 2024, from https://lavaan.ugent.be/tutorial/