R: Linear Mixed Effects Model (Längsschnittdaten)
– nlme Package und lme4 Package
Arndt Regorz, Dipl. Kfm. & MSc. Psychologie, 26.04.2023
Sie möchten Längsschnittdaten mit einem Linear Mixed Effects Model (LMEM, Mehrebenenmodell, Hiearchisches lineares Modell, linear growth model) auswerten? Dieses Tutorial zeigt Ihnen, wie Sie longitudinale Daten mit den R Packages nlme oder lme4 auswerten können.
Das hier vorgestellte Modell ist aus dem Grundlagenwerk von Hox et a. (2017) zur Mehrebenanalyse, dort in Kapitel 5 vorgestellt. Die Daten dazu können Sie auf der Internetseite von Hox herunterladen (http://joophox.net/mlbook2/DataExchange.zip), so dass Sie alle Auswertungen selbst nachvollziehen können. Es handelt sich hier um Längsschnittdaten von 200 Studierenden. Die Kriteriumsvariable "gpa" (Grade Point Average, Durchschnittsnote - hoher Wert = gute Note) wurde in sechs aufeinanderfolgenden Semester erhoben. Ebenfalls pro Semester wurde erhoben, wie viele Stunden die Studierenden neben dem Studium gearbeitet hattten (Variable "job"). Außerdem wurde das Geschlecht der Studierenden erfasst ("sex", 0 = männlich, 1 = weiblich) und die Durchschnittsnote während der Oberschule ("highgpa" = high school GPA).
INHALT
- Grundlagen
- Nullmodell
- Zeit als Prädiktor
- Time varying covariates
- Time invariant covariates
- Random Slope für die Zeit
- Cross-Level-Interaction
- Kovarianzstruktur für Level 1 Residuen
- Codierung der Variable Zeit
- Linear Mixed Effects Model für Experimente
- Modellierung nicht-linearer Effekte
- R-Code lme4
- Literatur
1. Grundlagen
Um das Maximum aus diesem Tutorial mitzunehmen, ist ein gewisse Grundverständnis der Mehrebenenanalyse/HLM hilfreich. Falls Sie dazu noch keine Basiskenntnisse haben, empfehle ich das Buch von Hox et al. (2017), insbesondere die Kapitel 2-5, 12-13.
Es gibt in R verschiedene Packages, um Mehrebenenmodelle zu schätzen. Am bekanntesten sind die beiden Packages lme4 und nlme. Das Modul lme4 ist das etwas moderne und ich würde ihm in Querschnittsdaten den Vorzug geben. Allerdings sieht das für ein Linear Mixed Effects Modell mit Längsschnittdaten anders aus.
In Modellen für Querschnittsdaten gibt es in der Regel die Annahme, dass die Level 1 Residuen nicht miteinander korreliert sind (Ausnahme im Querschnitt: Spatial Autocorrelation, wenn man im Raum benachbarte Objekte analysieren möchte, was in den Sozialwissenschaften selten vorkommt). Diese Annahme ist jedoch für Längsschnittdaten problematisch, insbesondere für intensive longitudinal data , wie sie z.B. in Tagebuchstudien (diary studies) vorkommen. Dort muss man nämlich davon ausgehen, dass es durchaus eine Korrelation zwischen benachbarten Messzeitpunkten gibt: Wenn z.B. drei Mal am Tag eine Messung erfolgt, dann kann ein Störeinfluss auf einen Messzeitpunkt (z.B. ein Konflikt im Büro) regelmäßig auch Auswirkungen auf benachbarte Messzeitpunkte (MZP) haben, was dann zu einer Autokorrelation der Residen führt.
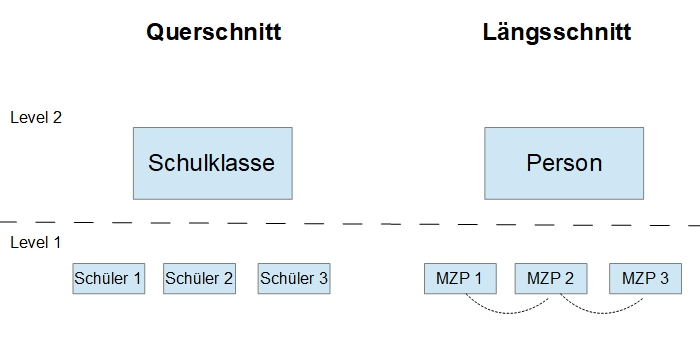
Für Längsschnittmodelle mit längeren Zeitintervallen ist das weniger ein Problem. Wenn Sie also beispielsweise für eine Entwicklungsstudie einmal pro Jahr die Daten messen, werden Sie häufig auch mit einem Modell ohne korrelierte Level 1 Residuen akzeptable Ergebnisse erzielen. Wenn Sie jedoch z.B. eine Tagebuchstudie (diary study) mit R auswerten wollen, dann müssen Sie unbedingt darauf achten, dass Sie auch die Level 1 Korrelationen modellieren, um keine verzerrten Schätzungen zu bekommen. Das kann zum Erstellungszeitpunkt des Tutorials jedoch nur das Package nlme und nicht das Package lme4.
Zwar ist der hier verwendete Beispieldatensatz ein Datensatz mit längeren Messintervallen, so dass auch eine Modellierung ohne Level 1 Korrelationen möglich ist. Damit Sie jedoch mit diesem Tutorial grundsätzlich in der Lage sind, Längsschnittdaten auszuwerten, werde ich Ihnen hier primär die Verwendung vom Package nlme demonstrieren, weil man damit insoweit für Längsschnittmodelle flexibler ist. Am Ende des Tutorials finden Sie jedoch den gleichen Code auch noch einmal für das Package lme4, falls Sie dieses bevorzugen.
Hinweis: Bei der folgenden Modellierung wurde aus didaktischen Gründen entsprechend dem Beispiel aus dem Buch von Hox et al. (2017) durchweg die ML-Schätzung statt der REML-Schätzung verwendet.
2. Nullmodell
Nach Laden der relevanten Packages beginnen wir mit der Schätzung eines Nullmodells, also noch ohne Prädiktoren. Sowohl für den Fixed Effect (gpa ~1) also auch für den Random Effect (~1|student) wird nur der Intercept geschätzt. Damit können wir z.B. die Intraklassenkorrelation berechnen.
# Längsschnittanalyse mit nlme-Package / lme-Funktion
library(nlme)
library(performance)
head(gpa2long)
# Nullmodell
nullmodell <- lme(gpa ~ 1, data=gpa2long, random= ~1|student, method="ML")
summary(nullmodell)

Oben im Output kann man Angaben zum Model fit ablesen. Neben zwei Fit-Indizes, AIC und BIC (hier sind jeweils kleinere Werte besser), wird die LogLikelihood ausgewiesen, die ein Maß für die Passungsgüte des gewählten Modells ist. Häufiger liest man dafür jedoch das Maß der Deviance - diese ist die LogLikelihood multipliziert mit -2, hier also (-456.7281) * (-2) = 913.5. Eine betragsmäßig kleinere LogLikelihood ist besser - wir werden im Folgenden noch sehen, in welchem Maß mit der Aufnahme von Prädiktoren sich die Modellgüte und damit die LogLikelihood verbessert.
Da wir noch keine Prädiktoren im Modell aufgenommen haben, bestehen die Fixed Effects nur aus der Schätzung des Intercepts.
Bei den Random Effects werden zwei aufgeführt: Der Random Intercept, also das Ausmaß der Schwankung des Intercepts (und hier ohne Prädiktoren daher des Mittelwerts) der Uninote zwischen den Studierenden, und das Level 1 Residuum, also die Notenschwankung innerhalb einer Person.
Hier ist wichtig zu wissen, dass das nlme-Package hier Standardabweichungen ausweist und keine Varianzen. Wenn Sie die Varianz berichten wollen (was eher Standard ist), dann müssen Sie diese Werte jeweils noch quadrieren.
Aus diesen beiden Werten kann man jetzt die Intraklassenkorrelation berechnen, wobei es dafür auch eine explizite Funktion gibt, die diese Arbeit abnimmt.
Aufruf der Intraklassenkorrelation (ICC):
icc(nullmodell) # mit library performance
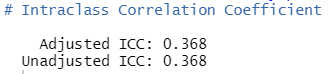
Wir haben im Beispiel also eine ICC von .37.
3. Zeit als Prädiktor
Jetzt ergänzen wir unser Modell um Zeit (occas) als ersten Prädiktor (gpa ~ 1 + occas).
# Modell mit Messzeitpunkt
messzeitpunkt <- lme(gpa ~ 1 + occas, data=gpa2long, random= ~1|student,
method="ML")
summary(messzeitpunkt)
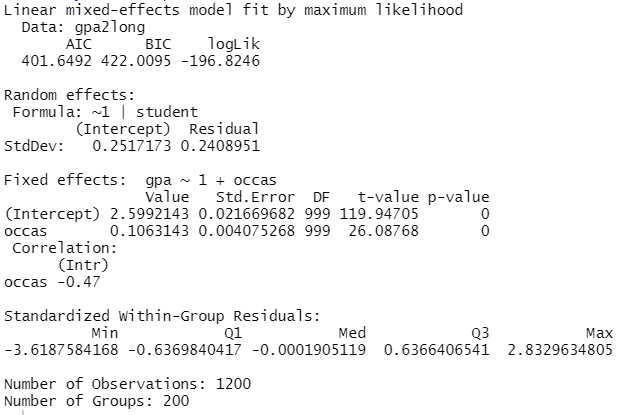
Zunächst sehen wir an der LogLikelihood, dass dieses Modell mit Zeit deutlich besser zu den Daten passt als das Nullmodell (-196.8 gegenüber vorher -456.7). Das werden wir auch in den weitern Schritten sehen - am Ende des Tutorials werden wir noch eine Übersicht über die Entwicklung des Modellfit betrachten.
Für die Zeit (occas) zeigt sich jetzt bei den Fixed Effects ein signifikant positiver Effekt - mit längerem Studium werden die Noten besser.
4. Time varying covariates
Neben der Zeit möchten wir in der Regel noch weitere Prädiktoren in unser Modell aufnehmen, sog. covariates (Kovariaten). Beim Linear Mixed Effects Model unterscheidet man dabei zwei Arten von Kovariaten: time varying und time invariant. Time varying Kovariaten sind Prädiktoren auf Level 1, die also zu jedem Messzeitpunkt einen anderen Wert annehmen können. In unserem Beispiel ist das die Variable Job Status (job).
# Modell mit time varying covariate (Job Status)
jobstatus <- lme(gpa ~ 1 + occas + job, data=gpa2long, random= ~1|student,
method="ML")
summary(jobstatus)
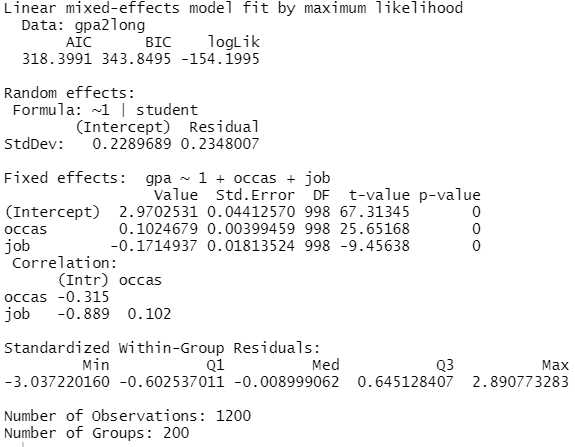
Der Nebenjob (job) hat einen signifikant negativen Effekt auf die Noten - je mehr Stunden jemand neben dem Studium arbeitet, desto schlechter die Noten.
5. Time invariant covariates
Im nächsten Schritt ergänzen wir das Modell um time invariant covariates. Das sind Prädiktoren auf Level 2, also Variablen, die für alle Messzeitpunkte gleich sind. Im Beispiel sind das die Abschlussnote der Schule (highgpa) und das Geschlecht (sex).
# Modell mit time invariant covariates (Schulnote, Geschlecht)
note_geschlecht <- lme(gpa ~ 1 + occas + job + highgpa + sex, data=gpa2long,
random= ~1|student, method="ML")
summary(note_geschlecht)
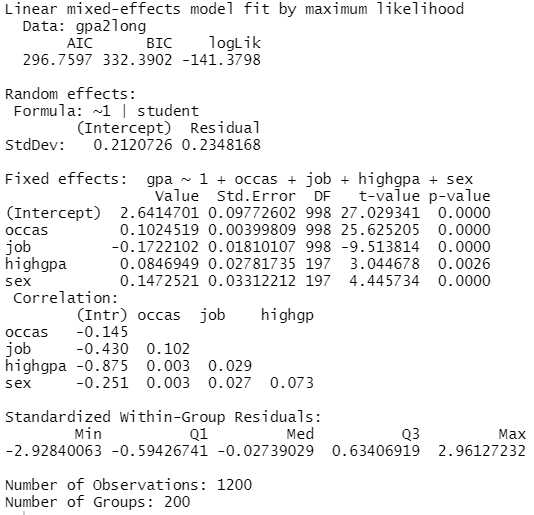
Bei den time invariant covariates sehen wir, dass Studierende mit besserer Schulnote auch im Studium bessere Noten haben (wenig überraschend). Außerdem sehen wir einen signifikanten Geschlechtseffekt - Studentinnen (sex = 1) haben bessere Noten als Studenten (sex = 0).
6. Random Slope für die Zeit
In einem weiteren Schritt führen wir jetzte eine Random Slope in das Modell ein, und zwar für den Messzeitpunkt. Das bedeutet wir lassen jetzt zu, dass der Effekt der Zeit sich zwischen verschiedenen Versuchspersonen unterscheidet. Das ist in der Komponente Random im Code enthalten (~1 + occas|student)
# Modell mit Random Slope für Messzeitpunkt
random_occas <- lme(gpa ~ 1 + occas + job + highgpa + sex, data=gpa2long,
random= ~1 + occas|student, method="ML")
summary(random_occas)
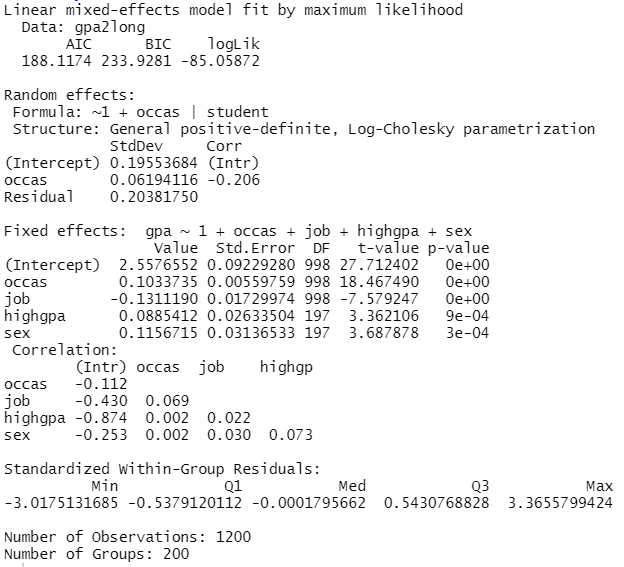
Jetzt sehen wir, dass bei den Random Effects zwei Effekte dazu gekommen sind. Zum einen gibt es jetzt einen Random Effect für occasion - das ist die Schwankung des Zeiteffektes zwischen den verschiedenen Studierenden. Und da wir mehr als einen Random Effect haben (Random Intercept und Random Slope für occasion) wird auch noch eine Korrelation dieser beiden Zufallseffekte ausgewiesen. Signifikanztests für Zufallseffekte sind nicht ganz trivial, siehe dazu das Tutorial p-Values Random Effects ermitteln.
7. Cross Level Interaction
Da wir im vorherigen Schritt eine nennenswerte Varianz in der Slope für die Zeit hatten, stellt sich die Frage, wodurch das u.U. zu erklären ist. Dafür kommen Cross-Level-Interaktionen in Frage, also hier Interaktionen von time invariant covariates mit der Zeit. In diesem Modell wollen wir prüfen, ob der Effekt der Zeit sich vielleicht zwischen den Geschlechtern unterscheidet (durch die Position occas:sex in den Fixed effects).
# Modell mit Cross-Level-Interaktion
cross_level <- lme(gpa ~ 1 + occas + job + highgpa + sex + occas:sex,
data=gpa2long, random= ~1 + occas|student, method="ML")
summary(cross_level)
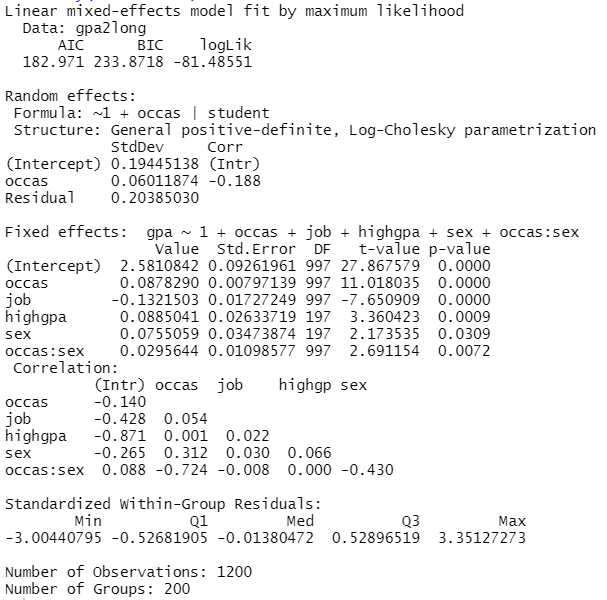
Die Cross-Level-Interaktion wird hier signifikant. Aufgrund des positiven Vorzeichens sehen wir, dass bei Studentinnen (sex = 1) der Zeiteffekt signifikant stärker ist als bei Studenten (sex = 0).
8. Kovarianzstruktur für Level 1 Residuen
Bisher hatten wir keine Kovarianzstruktur auf Level 1 vorgegeben. Die implizite Annahme war also, dass die Level 1 Residuen der Messzeitpunkte einer Person voneinander unabhängig sind.
Wenn man diese Annahme nicht aufrecht erhält (insbesondere bei intensiven Längsschnittdaten, ILD), kann man mit nlme auch eine Kovarianzstruktur der Level 1 Residuen schätzen - wenn man das nicht tut, wird diese vom Programm als unkorreliert angenommen. Im folgenden Code wurde eine der verschiedenen Kovarianzstrukturen angenommen, nämlich Autokorrelation ersten Grades
Autokorrelation ersten Grades bezieht sich auf die Korrelation zwischen einer Zeitreihe und ihrer eigenen um einen Lag (Zeitversatz) von einem Schritt verschobenen Version. Mit anderen Worten, es misst die Stärke der Beziehung zwischen einem Wert und dem vorherigen Wert in einer Zeitreihe.
Im Code ist das mit dem zusätzlichen Parameter correlation = corAR1(form = ~ 1|student) enthalten.
# Modell mit Level 1 Korrelationsstruktur (AR1)
correl_resid <- lme(gpa ~ 1 + occas + job + highgpa + sex + occas:sex,
data=gpa2long, random= ~1 + occas|student, method="ML",
correlation = corAR1(form = ~ 1|student))
summary(correl_resid)
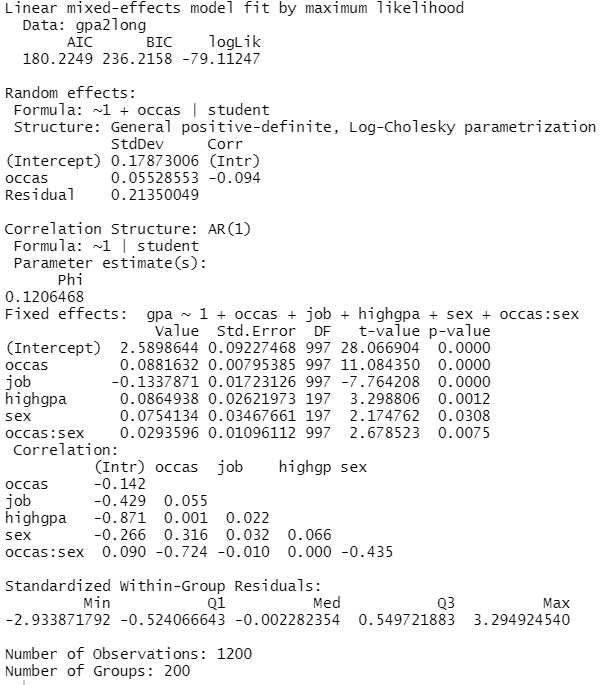
Beim Modellfit sieht man, dass sich das Modell nicht sehr stark verbessert (AIC wird noch geringfügig besser, BIC jedoch schlechter mit der Aufnahme der Autokorrelation). Das ist insoweit nicht erstaunlich, da der Zeitabstand zwischen den Messungen (ein Semester) relativ groß ist, so dass keine große Korrelation zwischen einzelnen Beobachtungen bzw. deren Residuen zu erwarten war. Auch bei den Parameterschätzungen sehen wir kaum Unterschiede zum vorherigen Modell.
Neu aufgenommen in den Output wurde jetzt die Parameterschätzung für die Korrelationsstruktur, hier die Schätzung für die Autokorrelation.
Diese Parametrisierung mit corAR1(form = ~ 1|student) setzt allerdings voraus, dass Daten für alle Personen und Zeitpunkte vorliegen, alle Zeitabstände gleich sind und die Daten auch innerhalb der Personen nach den Zeitpunkten geordnet sind. Denn das Programm muss ja wissen, welche Beobachtungen benachbart zueinander sind, um die Autokorrelation zu schätzen. In diesem Fall wird das aufgrund der Nachbarschaft im Datensatz getan.
Wenn mindetens eine dieser Bedingungen nicht gegeben ist, dann führt dieser Code zu einer fehlerhaften Schätzung. Wenn z.B. bei einer Person der Zeitpunkt 2 fehlt, dann würde das Modell die Zeitpunkte 1 und 3 für diese Person als direkt benachbart annehmen, was falsch ist. Für diesen Fall kann man bei der Autokorrelation auch eine Zeitvariable angeben, die dann für die Korrelationsschätzung herangezogen wird:
correlation = corAR1(form = ~ occas|student)
Es gibt aber neben der Autokorrelation 1. Grades noch andere mögliche Korrelationsstrukturen (einige zeitlich, einige auch räumlich/spatial); eine Liste kann man hier anfordern:
# Verschiedene Korrelationsstrukturen der Level 1 Residuen:
?corStruct

Zum Abschluss fordern wir hier noch eine Übersicht über die Gesamtergebnisse der verschiedenen Schätzungen an:
# Übersicht Gesamtergebnisse
anova(nullmodell, messzeitpunkt, jobstatus, note_geschlecht, random_occas,
cross_level, correl_resid)
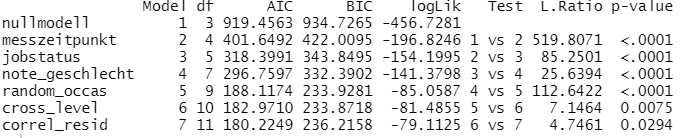
Zur Ermittlung der Likelihood-Ratio ("L.Ratio", vorletzte Spalte) werden vom System die LogLikelihood-Werte zweier benachbarter Modelle in die jeweilige Deviance umgerechnet (also * (-2)) und anschließend deren Differenz gebildet. Der sich daraus ergebende p-Wert in der letzten Spalte zeigt an, ob ein Modell signfikant besser zu den Daten passt als das Modell in der vorherigen Zeile.
9. Kodierung der Variable Zeit
Eine Schlüssefrage für ein Längsschnittmodell ist die Kodierung der Variable Zeit (bzw. der Variable Messzeitpunkt).
Zum einen kann man hinsichtlich der Zeit zwischen zwei verschiedenen Designs unterscheiden: Fixed Occasions und Varying Occasions.
Bei Fixed Occasions werden bei den verschiedenen Untersuchungseinheiten / Versuchspersonen die Messungen zum gleichen Zeitpunkt durchgeführt. Das o.g. Beispiel ist ein Modell mit Fixed Occasions (alle zum 1., 2., 3. usw. Semester).
Bei Varying Occasions ist der Zeitpunkt der Messung zwischen den Untersuchungseinheiten / Versuchspersonen unterschiedlich. Ein Beispiel dafür wäre eine Entwicklungsstudie, die im Klassenkontext erhoben wird. Wenn zu Beginn der 5. Klasse Daten erhoben werden, sind einige Schülerinnen und Schüler vielleicht 10.4 Jahre alt und andere 10.9 Jahre. In diesem Fall würde man das, variable, Alter als Zeitvariable nutzen und nicht die Klassenstufe, weil in der Kindheitsentwicklung ein halbe Jahr schon einen sehr deutlichen Effekt haben kann.Eine zweite Frage im Zusammenhang mit der Zeit ist der Nullpunkt der Variable. Die Schätzung von Intercepts ergibt jeweils den vorhergesagten Wert der Kriteriumsvariable, wenn die Prädiktoren eine Ausprägung von Null haben. Daraus ergibt sich, dass es beispielsweise nicht sinnvoll ist, Jahreszahlen für die Variable Zeit zu verwenden. Wenn Sie eine Untersuchung der Jahre 2018-2022 vorgenommen haben und die Jahreszahl als Variable verwenden, dann würde der Intercept die Schätzung der Kriteriumsvariable zum Jahre 0, also vor 2000 Jahren, angeben. Das ist relativ selten ein für uns relevanter Wert.
Daher bietet es sich häufig an, die Zeit so umzucodieren, dass der erste Messzeitpunkt den Wert von 0 bekommt. Statt 2018-2022 würde man die Zeit also als 0-4 codieren und der Intercept gibt dann die Verhältnisse zu Beginn der Messung (im Jahr 2018) wieder.
Wenn hingegen das Alter als Zeitvariable verwendet wird, ist es meistens sinnvoll, diese zu zentrieren. Denn die unzentrierte Altersvariable würde dazu führen, dass der Intercept den Effekt bei einem Alter von Null Jahren ausweist.
10. Linear Mixed Effects Model für Experimente
Linear Mixed Effects Modelle können auch gut für die Auswertung von Verlaufsdaten im Rahmen eines Experiments genutzt werden. Dabei ist die Zugehörigkeit zur Experimental- oder Kontrollgruppe eine zeitinvariante Kovariate auf Level 2.
Der für ein Experiment entscheidene Effekt ist dann die Cross-Level-Interaktion zwischen der Zeit (Level 1) und der Gruppenzugehörigkeit (Level 2). Wenn diese signifikant wird, weiß man, dass sich der Zeitverlauf der abhängigen Variable zwischen den beiden Gruppen unterscheidet - und genau das möchte man bei einem Experiment in der Regel wissen.
11. Modellierung nicht-linearer Effekte
Mit einem derartigen Modell können Sie auch relativ einfach nicht-lineare Effekte der Zeit modellieren. Vielleicht ist es für Ihre Daten keine realistische Annahme, dass die Kriteriumsvariable sich im Zeitverlauf linear verändert? Sie können auch u-förmige, umgekehrt u-förmige oder sonstig gebogene Zeitverläufe der Daten modellieren.
Dazu schließen Sie einfach neben der Zeit auch noch zusätzlich das Quadrat der Zeit in Ihr Modell als Fixed Effekt ein. In unserem Beispiel hier würden wir also eine neue Variable anlegen, occas2 = occas * occas, und diese zusätzlich in das Modell einschließen.
12. R-Code für Linear Mixed Effects Model mit lme4 Package
Aus den genannten Gründen ist für Längsschnittdaten das Package nlme die flexiblere Lösung. Wenn Sie jedoch für Längsschnittdaten mit relativ langen Messintervallen lieber lme4 als Package verwenden möchten, finden Sie hier den R-Code:
library(lme4)
library(lmerTest)
library(performance)
head(gpa2long)
# 1. Nullmodell
nullmodell <- lmer(gpa ~ 1 + (1|student), data=gpa2long, REML = FALSE)
summary(nullmodell)
icc(nullmodell) # mit library performance
# 2. Modell mit Messzeitpunkt
messzeitpunkt <- lmer(gpa ~ 1 + occas + (1|student), data=gpa2long,
REML = FALSE)
summary(messzeitpunkt)
# 3. Modell mit time varying covariate (Job Status)
jobstatus <- lmer(gpa ~ 1 + occas + job + (1|student), data=gpa2long,
REML = FALSE)
summary(jobstatus)
# 4. Modell mit time invariant covariates (Schulnote, Geschlecht)
note_geschlecht <- lmer(gpa ~ 1 + occas + job + highgpa + sex + (1|student),
data=gpa2long, REML = FALSE)
summary(note_geschlecht)
# 5. Modell mit Random Slope für Messzeitpunkt
random_occas <- lmer(gpa ~ 1 + occas + job + highgpa + sex +
(1 + occas|student), data=gpa2long, REML = FALSE)
summary(random_occas)
# 6. Modell mit Cross-Level-Interaktion
cross_level <- lmer(gpa ~ 1 + occas + job + highgpa + sex + occas:sex +
(1 + occas|student), data=gpa2long, REML = FALSE)
summary(cross_level)
13. Literatur
Hox, J. J., Moerbeek, M., & Van de Schoot, R. (2017). Multilevel analysis: Techniques and applications (3rd edition). Routledge.
Weitere Tutorials zu Linear Mixed Effects Models / Mehrebenenmodellen:
- Mehrebenenanalyse (mit R) 1: Grundlagen mit lme4 Package
- Mehrebenenanalyse (mit R) 2: Voraussetzungen prüfen
- Mehrebenenanalyse (mit R) 3: Zentrierung
- Mehrebenenanalyse (mit R) 4: p-Values Random Effects ermitteln
- Mehrebenenanalyse (mit R) 5: Ergebnisse berichten
- Mehrebenenanalyse (mit R) 6: Robuste Schätzung
- Mehrebenenanalyse (mit R) 7: Grundlagen mit nlme Package